INTRODUCTION
A형간염은 A형간염 바이러스(Hepatitis A virus, HAV)에 오염된 음식이나 물을 섭취하거나 감염자와의 접촉 등으로 전파되는 급성 간염 질환으로 지역의 사회경제적 여건에 따라 다양한 양상을 보인다 (1). 국내의 경우 위생수준이 낮았던 60~70년대에는 소아기 감염이 주로 나타났지만 최근에는 개인위생 수준의 향상과 생활환경 개선, 예방접종의 도입으로 소아기 감염은 거의 없어졌다. 대신 항체 양성률이 낮은 20~40대 성인 연령층에서의 현증 A형간염 사례가 증가하고 있으며, 이러한 역학적 변화에 따라 사회경제적 활동이 가장 많은 연령층의 감염으로 인한 비용 손실도 증가하고 있는 상황이다 (2).
국내 A형간염 관리는 2011년부터 표본감시체계에서 전수감시체계로 변경되어 운영하고 있으며, 질병관리청 전수감시 감염병 통계에 따른 A형간염 발생 신고는 2011년 5,521명이었고, 이후 약 1천여명 수준으로 신고되었다. 2016~2017년 4,500여명 내외로 급증했으나 2018년 2,437명으로 감소하였고, 2019년 오염된 조개젓 섭취로 인한 대규모 유행으로 전국적으로 17,000여명이 발생하였다 (3, 4). A형간염 발생이 많은 경기, 인천, 충남 등에 비해 부산에서의 발생 건수는 적은 편이나 2016년에는 생굴 추정, 2019년 조개젓으로 인한 집단 발생이 있었으며, 일반 수인성 질환과 마찬가지로 단체 급식이나 외식 증가로 인한 집단발병 가능성이 증가하고 있다 (5).
HAV는 단일 혈청이지만 유전자 염기서열 변이에 따라 I부터 VII까지 7가지 유전자형으로 분류되고 I형과 III형은 다시 A와 B subtype으로 나뉘며, 7가지 유전자형 중 사람에게 감염을 일으키는 유전자형은 4개(I, II, III, VII)로 알려져 있다 (1). 또한 HAV는 RNA 바이러스이지만 염기서열 변화가 적고 지역별로 토착화된 유전자형을 나타내므로 유전자형 분석과 같은 분자역학적 감시를 통해 전파경로 파악이 가능하다 (1).
하지만 현재 의료기관에서 개별적으로 발생하는 A형간염의 경우 항체검출검사(IgM)로 확진하고 있어 유전자형 분석은 이루어지지 않고 있다. 그리고 수인성 및 식품매개 감염병 관리지침의 수인성・식품매개 감염병 유행 역학조사 기준에 따라 2건 이상의 사례가 역학적인 연관이 있다고 판단되는 경우에만 관할 보건환경연구원에서 A형간염 바이러스 유전자 검출검사를 실시하고 유전자형을 분석하여 집단환자 발생의 원인 규명에 활용하고 있다. 이러한 이유로 A형간염에 관한 연구는 항체 양성률이나 임상 특성 등에 관한 연구가 대부분으로 유전자형과 관련한 연구는 검체 수집이 가능한 일부 의료기관이나 연구원에서 이루어지고 있으며, 특히 A형간염 발생이 많은 경기, 인천, 충남 등 타지역에 비해 발생 건수가 적은 부산 지역의 A형간염 유전자형에 대한 연구는 부족한 실정이다.
따라서 본 연구에서는 수인성・식품매개질환이지만 질병관리청 감시사업의 대상병원체에서 제외되어 있는 A형간염의 검출 현황을 조사하여 A형간염의 조기 인지를 위한 대상병원체 추가 여부를 파악하고자 하였다. 또한 2022년 부산지역 의료기관에서 A형간염으로 신고된 검체에 대한 HAV 특이 유전자 검사와 염기서열 분석으로 부산지역에서 유행하는 HAV 유전자형을 알아보고자 하였다.
MATERIALS AND METHODS
연구대상
수인성・식품매개질환 실험실 감시사업 검체 중 A형간염 감염 실태 조사를 위해 2019년 1월부터 2022년 10월까지 부산지역 협력병원으로부터 의뢰된 대변 검체 1,793건을 대상으로 HAV 특이유전자 검출 검사를 실시하였다. 또한 부산광역시 건강정책과와 감염병관리지원단의 협조로 2022년 4월부터 11월까지 부산지역 의료기관에서 IgM 양성으로 신고된 A형간염 환자 및 병원체보유자 잔여 혈청 12건을 수집하여 HAV 특이유전자 검출 여부를 확인하였다.
HAV RNA 추출
대변 검체는 phosphate buffered saline으로 10%의 부유액을 제조하여 강하게 진탕하고 원심분리(3,000 rpm, 20분)한 상등액을 사용하였고, 혈청은 별도의 전처리 없이 추출에 사용하였다. 검체 200 µL를 취하여 핵산추출기(Genolution, Korea)를 이용하여 viral RNA를 추출하였다.
HAV 특이유전자 검사
HAV 특이유전자 검사는 VP3-VP1 부위를 타겟으로 하여 Table 1의 primer와 Table 2의 반응 조건으로 Applied Biosystem PCR (Veriti, USA)를 이용하여 증폭하였다. 1차 RT-PCR, 2차 nested PCR 실시 후 QIAXcel Advanced System (Qiagen Inc. CA, USA)을 이용하여 HAV RNA임을 확인하였다.
Table 1.
The sequence of primers for RT-PCR and nested PCR to detect HAV VP3-VP1 region
Table 2.
PCR reaction conditions for detection of Hepatitis A virus gene
1차 RT-PCR은 HyQTM Onestep RT-PCR Premix kit (SNC, Korea)를 사용하여 premix 13.5 µL에 10 pmole의 HAV1 forward 및 reverse primer 각 1 µL씩, purified RNA 5 µL, RNase-free water 4.5 µL를 넣은 후 최종 용량이 25 µL가 되도록 하였다. Nested PCR은 HyQTM PCR premix kit (SNC, Korea)를 사용하여 master mix 10 µL에 10 pmole의 HAV2 forward 및 reverse primer 각 1 µL씩, 1차 RT-PCR product 1 µL, RNase-free water 7 µL를 넣은 후 최종 용량이 20 µL가 되도록 하였다.
A형간염 바이러스 유전자형 확인 및 계통분석
A형간염 바이러스 PCR 검사 양성검체를 대상으로 VP3-VP1 junction (186 bp) 부위를 증폭하였으며 증폭산물은 시퀀싱을 위해 (주)제노텍(대전, 한국)에 의뢰하였다. 분석된 염기서열을 NCBI (National Center for Biotechnology Information)의 BLAST search program을 이용하여 기존에 보고된 바이러스와의 상동성을 확인하였으며, 이러한 염기서열을 토대로 계통분석을 실시하였다. 검출된 A형간염 바이러스의 염기서열은 Genbank에 등록된 A형간염 바이러스주들과 비교하였으며(Table 3), MEGA11 프로그램을 사용하여 phylogenetic tree를 작성하였다.
Table 3.
Hepatitis A virus strains from GenBank used in the phylogenetic analysis described in this study
RESULTS AND DISCUSSION
수인성・식품매개질환 병원체 감시사업에서의 A형간염 감염 실태 결과
2019년 1월부터 2022년 10월까지 부산지역 수인성・식품매개질환 실험실 감시사업 협력병원에서 의뢰한 대변검체 총 1,793건(2019년 669건, 2020년 349건, 2021년 337건, 2022년 438건)에 대한 HAV PCR 검사 결과 2019년 2월 검체 1건에서 검출되었다.
질병관리청 주관으로 수행하는 수인성・식품매개질환 실험실 감시사업은 각 지역의 협력병원에 내원한 설사, 구토, 복통 증상을 나타내는 환자 대변 검체에서 원인 병원체를 조기 발견하고 유행을 예측하기 위한 사업으로 대상병원체는 세균 10개속, 바이러스 5종(노로, 로타, 아스트로, 사포, 장아데노), 원충 4종이며 A형간염 바이러스는 제외되어 있다. A형간염의 임상 증상 중에도 설사나 구토 등이 포함되므로 감시사업 검체에서 검출이 가능할 것으로 생각되었으나 검출률 0.06%로 대상 병원체에 A형간염을 추가하여 추진하기에는 효율성이 낮은 것으로 판단되었다. 검출된 1건은 2019년 8월 조개젓으로 인한 집단발생 이전인 2월에 의뢰된 검체로 다른 감시사업 대상병원체는 모두 검출되지 않았다. 본 연구와 마찬가지로 울산보건환경연구원에서도 수인성・식품매개질환 실험실 감시사업 검체 및 잠복결핵 의심환자 검체 등 700건을 대상으로 A형간염 바이러스 유전자 검사를 실시하였지만 모두 불검출이었다 (6).
개별사례 검체에서 HAV RNA 검사 결과
2022년 4월부터 11월까지 부산지역에서 IgM 양성으로 신고된 A형간염 환자는 40명으로, 이 중 의료기관의 협조로 검체를 수집할 수 있었던 혈청 12건 중 5건에서 HAV 특이유전자가 검출되어 검출률은 41.7% 였다.
혈청에서 HAV 특이유전자 검출을 실시한 기존 보고에서 58.0~81.7%의 검출률을 나타낸 것에 비해 본 연구에서의 검출률은 41.7%로 낮았는데, 검체 보관량이 부족하여 실험이 불가한 경우도 있었으며, 증상은 없으나 건강검진이나 수술을 위한 입원 전 검사에서 IgM 양성으로 신고된 경우 혈액 내 바이러스 농도가 낮아 PCR에서 검출되기 어려웠던 것으로 보인다 (7, 8, 9).
유전자형 확인 및 계통분석
A형간염 바이러스 PCR 검사에서 양성이었던 2019년 2월 감시사업 검체 1건과 2022년 개별사례 검체 5건에 대한 염기서열 결과로 유전자형을 확인한 결과 Table 4와 같이 2019년 감시사업 검체 1건은 IB형, 2022년 개별사례 검체 5건 중 3건은 IA형, 2건은 IIIA형이었다. IA형 중 22-6과 22-9는 2006년 국내에서 분리된 EU049560과 각각 95%와 97%의 유사도를 나타내었고, 22-12는 2005년 국내에서 분리된 EU049550과 96% 유사도를 나타내었다. IB형으로 확인된 감시사업 검체 19-02-42는 2008년 중국의 인체 검체에서 유래한 KF006844과 유사도 94%였으며, 표에 나타내지는 않았지만 국내 지표수에서 2009년에 분리한 HF679554와도 94%의 유사도를 나타내었다. IIIA형인 22-4와 22-7은 2008년 국내에서 분리된 FJ793268과의 유사도가 각각 95%, 98%이었다. 또한 본 연구에서 검출한 6건 외에 2019년 부산에서 발생한 A형간염 사례로부터 유전자형이 IA형임을 확인하였던 6건을 추가한 총 12건의 계통분석 결과는 Fig. 1과 같다. 추가한 6건 중 2019년 8월 조개젓으로 인한 집단발생 사례 4건은 대표 인체검체 3건(19-11, 19-62, 19-547)과 식품검체 1건(19-Food)이고, 나머지 2건은 집단발생 이후 2019년 11월 심층역학조사를 위해 개별 사례로 의뢰되었던 인체검체(19-1299, 19-1669)이다.
Table 4.
General characteristics and genetic analysis of 6 cases in this study
|
Sample name | Genotype | Case |
Date of sample collection | Sources |
Nearest NCBI BLAST Accession No. (Similarity) |
|---|---|---|---|---|---|
| 19-02-42 | IB | Enter-Net* | 2019.02 | Stool | KF006844 (94%) |
| 22-4 | IIIA | Sporadic | 2022.03 | Serum | FJ793268 (95%) |
| 22-6 | IA | Sporadic | 2022.06 | Serum | EU049560 (95%) |
| 22-7 | IIIA | Sporadic | 2022.06 | Serum | FJ793268 (98%) |
| 22-9 | IA | Sporadic | 2022.07 | Serum | EU049560 (97%) |
| 22-12 | IA | Sporadic | 2022.11 | Serum | EU049550 (96%) |
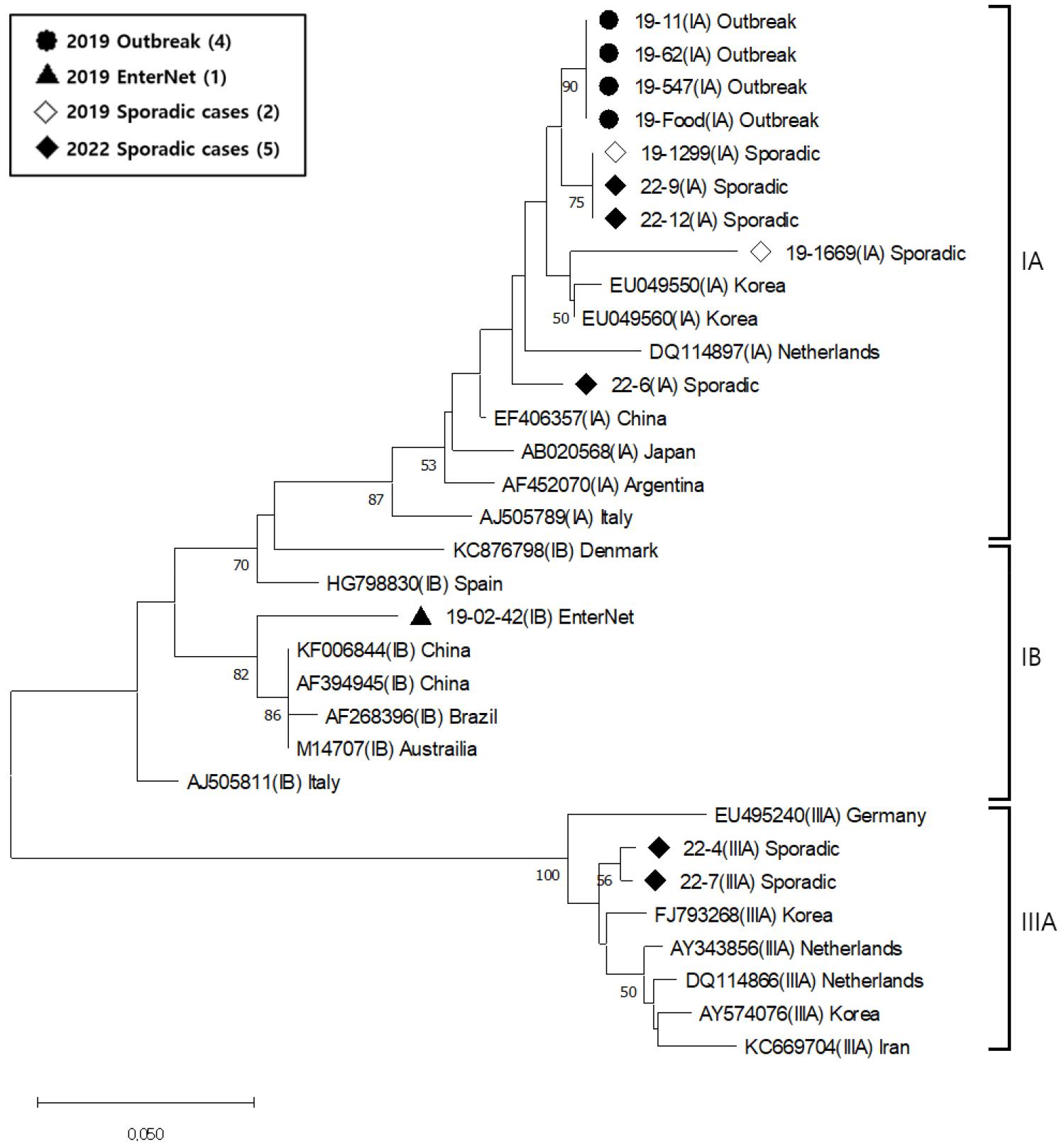
Fig. 1
Phylogenetic analysis of the HAV genotype based on the VP3-VP1 region. The tree was constructed using Neighbor- joining method with 1,000 bootstrap replicates by MEGA 11.
HAV 유전자형 중 전세계적으로 가장 많이 분포되어 있는 I형 유전자형의 경우 IA와 IB의 아형으로 나뉘고, IA형은 주로 북아메리카, 유럽, 중국, 일본과 태국 등에서, IB형은 지중해, 호주, 아프리카 등에서 발견된다 (10, 11). IIIA형의 경우 인도, 스리랑카, 네팔, 말레이시아와 미국에서 발견되는 등 유전자형에 따라 유행하는 지역적 분포 특성을 나타내고, HAV 유전자형 간의 염기서열 차이는 15~25%, 아유전자형 간에는 7.5%이고, 염기서열의 변화가 거의 없으므로 유전자형 분석과 염기서열 변화 확인은 A형간염의 유입 확인 등 역학적으로 의미가 있다고 할 수 있다 (1, 11). 국내에서 분자생물학적인 방법으로 HAV 유전자형이 처음 보고된 것은 2001년으로 이 연구에서 1997년과 1998년 A형간염 환자 18명에서 바이러스를 분리하여 HAV 유전자형이 IA였음을 확인하여 국내 토착형은 IA형으로 알려져 있었다 (7). 이후 2004년 국내 급성간염 환자에서 분리된 바이러스의 유전자형이 파키스탄의 IIIA형 분리주와 높은 상동성을 나타냈음이 보고되었으며, 이후 국내 집단 발병 사례에서 IB형과 IIIA형의 동시 유행이 보고되었다 (12, 13). 질병관리청에서 2005년부터 2010년까지 국내 A형간염 환자에서 검출한 HAV의 분자역학적 분석 결과 2005~2006년 IA형 83.3%, IIIA형 6.7%에서 점차 IIIA형 바이러스 검출 비율이 증가하여 2008년에는 IA형 48.6%, IIIA형 47.7%로 증가하였고, 2010년에는 IA형이 7.2%인데 반해 IIIA형이 92.8%로 증가하여 유행 양상이 변하였다고 보고하였다 (14). 또한 이러한 유행 양상의 변화는 A형간염 유행지역의 해외여행이나 외국인 근로자 또는 수입 식재료에 의해 유입되는 것으로 추측되고, 외국에서 새로운 HAV 유전자형의 유입 가능성을 시사하였으며 IA형과 IIIA형이 동시에 토착화되었다고 하였다 (14). 비슷한 시기에 지역별로 보고된 서울, 경기, 대전, 광주 및 전남 지역의 유전자형 연구에서도 IIIA형이 IA형보다 높은 비율로 유행하고 있다고 하였고, 서울의 경우 2002년 IA가 93.3%로 주를 이루었으나 약 10년 후인 2011년에는 IIIA가 69.2%를 차지하였다고 하였다 (8, 9, 15, 16, 17, (18). 이 시기 이후 국내 유행하는 A형간염 바이러스의 유전자형에 대한 연구는 집단발생으로 인한 사례와 2019년 조개젓으로 인한 집단 발생에 대한 사례가 보고되고 있으며 모두 IA형에 의한 것으로 보고되고 있다 (3, 4, 5, 19, 20). 본 연구에서는 2019년과 2022년 검체에서만 유전자형 확인이 가능하였으나, 국내에서 주로 유행하고 있는 I형과 III형이 모두 검출되었으며, 특히 I형의 경우 subtype인 IA와 IB가 모두 검출되어 부산지역에서도 다양한 유전자형이 유행하고 있음을 확인하였다.
A형간염의 발생 경로가 다양화됨에 따라 유전자형 유행에도 변화가 발생할 수 있으며, 특히 부산의 경우 공항과 항만을 모두 보유한 도시로 다른 지역에 비해 국외 유입의 가능성이 크다. 부산광역시는 A형간염의 전수감시 이후로 발생이 가장 많았던 2016년 A형간염의 선제적 예방관리와 전파차단을 위한 관리방안을 마련을 위해 2017년 ‘부산광역시 A형간염 관리지침’을 마련하고, 지역 내 발생하는 모든 A형간염 사례들에 대한 역학조사 후 밀접 접촉자를 대상으로 예방접종 사업을 추진하고 있다. 특히 2019년 조개젓으로 인한 집단 발생 이후 2020년에는 항체양성률이 낮은 20~40대의 만성 간질환자를 대상으로 한 예방접종 등 A형간염 관리를 위해 노력하고 있다. 이러한 노력의 일환으로 부산지역 내 의료기관과 보건환경연구원의 네트워크 구축으로 집단발생 사례 외에도 개별 사례에 대한 검체 수집과 실험실 분석으로 지역 내 유행하는 유전자형 분석 등 과학적인 자료의 지속적인 축적이 필요할 것으로 판단된다. 또한 기존 연구에서 유전자형에 따른 입원기간, 급성간부전, 사망의 발생은 차이가 없으나 IIIA형에 의한 감염이 IA형에 비해 alanine aminotransferase (ALT), aspartate aminotransferase (AST), 총 빌리루빈 수치가 더 높고 열과 전신 쇠약감의 증상이 조금 더 심한 양상을 나타낸다는 보고 (13, 16)가 있어 실험실 분석 연계 시 지역에서 발생하는 환자 임상정보와 유전자형과의 종합적인 데이터 분석도 가능할 것으로 생각된다.
CONCLUSION
사회, 환경적인 변화로 감염병의 유입과 유행도 변화하고 있으며, 감염병 발생 시 확산 차단을 위한 원인병원체의 검출과 유전자형 분석 등이 필요하나 현재 개별적으로 발생하는 A형간염은 진단을 위한 검사만 이루어져 병원체 특성 파악은 이루어지지 않고 있다. 본 연구에서 목적으로 하였던 급성설사질환 감시사업에서의 A형간염 조기 인지는 검출률이 낮아 효율성은 없었으나 의료기관의 협조로 수집한 개별사례 검체에서 유전자형 파악이 가능했다는 점에서 의료기관과 실험실간 네트워크 구축에 대한 검토의 계기가 될 것으로 기대한다. 또한 기존의 부산지역 A형간염 연구가 항체양성률에 대한 주제가 대부분이었으나 본 연구에서는 병원체의 유전자형에 대해 다루었다는 점에서 병원체 특성 연구에 대한 기초 자료로 활용될 것으로 생각되며, 지속적인 자료 수집을 통한 역학적인 변화 파악이 필요할 것으로 사료된다.




