INTRODUCTION
Carbapenem Resistant Enterobacteriaceae (CRE)는 carbapenem계 항생제에 내성을 나타내는 장내세균속균종이다. 특히 CPE (Carbapenemase Producing Enterobacteriaceae)는 carbapenemase를 생성하는 carbapenem계 항생제내성 장내세균속균종이며, 여러 다른 계열의 항생제에도 내성이 동시에 확인됨에 따라 치료용 항생제의 선택 압력(Antibiotics selective pressure)이 작용하는 것으로 알려져 있다 (1).
CPE를 포함하는 CRE 감염증의 치료는 colistin, tigecycline, gentamicin과 그 외 Carbapenem계 항생제의 병합요법 등이 사용된다 (2). 특히, World Health Organization (WHO)가 최후 수단의 항생제로 정한 colistin은 1940년 대에 개발되었으나, 신독성 등의 이상반응으로 사용하지 않았다. 하지만 다제내성균의 등장으로 인하여 마지막 선택 치료제로 다시 사용이 늘고있는 추세이다 (2). 이러한 colistin에 내성을 나타내는 CRE의 전파 사례는 이미 프랑스, 인도, 브라질(Klebsiella pneumoniae)과 한국(Enterobacter cloacae complex)에서 보고된 바 있다 (3, 4, 5, 6).
colistin은 Polymyxins 계열의 항생제로 Paenibacillus polymyxa균으로부터 분리되었고 A, B, C, D와 E group으로 나뉘며 polymyxin E (colistin)와 polymyxin B만 임상에 사용되고 있다 (7).
colistin의 작용 기작은 그람 음성균 외막의 lipopolysaccharides 와 phospholipids에 결합하여 세균 세포막 파괴로 세포를 사멸 시킨다. 이러한 colistin에 대한 세균의 내성 획득 기작은 크게 두 가지인데 첫 번째는 polysaccharide 구조변화를 통한 항생제 결합력 및 막 투과성 감소를 통해 이루어지며, 이때 구조 변화는 chromosomal mutation을 통하여 이루어지기 때문에 내성 유전자의 세균 간 수평 전달의 가능성은 낮다. 두 번째 내성 획득 기작은 plasmid에 의한 내성 유전자 전달이다. Mobile colistin resistance (mcr) gene은 cytoplasmic transmembrane 단백질인 MCR을 발현한다. 이 단백질은 phosphoethanolamine (pEtN) transferase으로 세균 세포 표면의 lipopolysaccharide의 lipid A 에 부착하여 음성인 세포막의 극성을 변화시켜 양성 극성의 colistin의 결합을 저해 시킨다. mcr 유전자는 plasmid 의해 동일 균종 뿐만 아니라 다른 균종 간 수평 전달이 가능하여 쉽고 빠르게 확산이 가능하여 내성균의 전파 차단에 있어 주요 관심대상이다 (8, 9, 10, 11). 현재까지 mcr-1~mcr-10 형까지 10종의 유전형이 알려져 있으며, 2000년대 이후 중국, 독일, 노르웨이등에서 강물, 생활하수 등의 환경과 닭고기, 채소 등 식품으로 부터 분리된 장내세균에서 mcr-1 을 비롯한 다양한 유전형이 확인되어 왔다 (12, 13, 14, 15).
따라서 본 연구에서는 한국의 강원도 지역내 의료기관으로 부터 수집된 CRE 균주를 대상으로 colistin 내성 현황 및 내성 균주의 분자유전학적 특성을 파악하고, 이들 균주 간 전파 여부를 pulsotype 분석을 통하여 확인하였다.
MATERIALS AND METHODS
검체수집 및 세균동정
2019~2021년 강원도의 42개 의료기관으로 부터 CRE 임상분리균주 666건을 수집하였다. 수집된 균주는 Carbapenem계 항생제 내성균 선별 배지인 mSuperCARBA ChromAgar (CHROMagar)에서 재분리 하고, 순수 분리된 균주는 Nutrient Agar (NA, Oxoid, UK)에 도말 하여 37℃에서 18~24시간 배양한 후 MALDI Biotyper (Bruker, Germany)와 Vitek2 Compact (BioMerieux, France)를 사용하여 확인 동정 하였다. MALDI Biotyper로는 직접 도말 하여 동정하였고, Vitek2 Compact로는 균을 0.5 McFarland (McF)로 희석하여 Gram Negative 카드(Biomerieux, France)를 사용하여 동정하였다.
항생제 감수성 검사
항생제 감수성 시험은 CLSI 및 EUCAST 의 기준을 참고하였으며, 총 25종의 항생제 중 20종 [Penicillins (ampicillin, piperacillin), ß-Lactamase inhibitors (amoxicillin-clavulanic acid, ampicillin-sulbactam), Cephems (cefazolin, cefoxitin, cefotaxime, ceftazidime, cefepime), Monobactams (aztreonam), Fluoroquinolones (ciprofloxacin), Folate pathway inhibitors (trimethoprime-sulfamethoxazole), Aminoglycosides (amikacin, gentamicin, tobramycin), Tetracyclines (tigecycline, tetracycline), Quinolones (nalidixic acid), Phenicoles (chloramphenicol), Nitrofurans (nitrofurantoin)]은 디스크확산법(Disk diffusion method)으로, 5종 Carbapenems (doripenem, imipenem, meropenem, ertapenem), Polymyxins (colistin)은 액체배지미량희석법 (Broth microdilution method)으로 항생제 감수성을 평가 하였다 (16, 17, 18). 디스크확산법(Disk diffusion method)은 분리된 균을 Nutrient Agar (NA, Oxoide, UK)에 도말 하고, 37℃에서 18시간 배양하여 자란 균을 0.85% saline 3 ㎖에 현탁 하여 탁도가 0.5 McF가 되도록 하였다. 준비된 균 현탁액을 RetroC80 (BioMerieux, France)을 이용하여 Muller-Hinton Agar (MHA, KisanBio, Korea) 배지에 골고루 도말 하고 Disc dispenser (Oxoide, UK)를 사용하여 항균제 디스크를 배지 표면에 부착시킨 후, 35℃에서 16~18시간 배양하였다. 항생제에 의한 억제대 지름을 측정하기 위해 BIOMIC V3 (Giles Scientific, USA)를 사용하였다.
액체배지미량희석법(Broth microdilution method)은 동정 된 균을 Nutrient Agar (NA, Oxoid, UK) 배지에 도말 하여 37℃에서 18시간 배양한 후 0.85% saline 3 ㎖에 현탁 하고 0.5 McF가 되도록 하였다. 준비된 균 현탁액 10 ㎕ 를 Muller Hinton Broth (MHB, Thermo, T3462) 11 ml에 잘 섞어준 후 carbapenem계 항생제 MIC (Minimum Inhibitory Concentration) plate (Sensititre Customized Gram-Negative Plate –KORN, TREK Diagnostic Systems), colistin MIC plate (Sensititre Customized Gram-Negative Plate –KNIHCOL, TREK Diagnostic Systems)의 각 well에 50 ㎕ 를 접종한 뒤 sealing tape을 붙이고 35±2℃에서 16~20시간 배양한 후 doripenem, imipenem, meropenem은 ≥4 ㎎/L, ertapenem은 ≥2 ㎎/L, colistin은 ≥4 ㎎/L 의 MIC 를 내성으로 판단하였다. 항생제 감수성 시험의 유효성 평가를 위하여 Escherichia coli ATCC 25922 를 사용하였다.
내성유전자 확인
주형 DNA는 순수 분리된 세균 집락을 증류수에 부유한 액을 가열하여 준비하였으며, 유전자 증폭은 MaximeTM PCR PreMix (i-StarTaq, Intron, Korea)와 각 표적 유전자에 따른 특이 primer를 사용하였다. 항생제 감수성 시험결과에 따라 Carbapenem계 항생제 4종 중 1가지 항생제라도 내성이 있는 균을 대상으로 carbapenemase 10종(IMP, Imipenemase; OXA, Oxacillinase; VIM, Verona Paulo metallo-ß-lactamase; NDM, New Delhi metallo-ß-lactamase; KPC, Klebsiella pneumoniae carbapenemase; GES, Guiana extended-spectrum ß-lactamase; SME, Serratia marcescens enzyme; GIM, German imipenemase; SIM, Seoul imipenemase; SPM, Sao ampicillinase) 유전자의 확인을 위한 PCR을 수행하였다 (19).
또한 colistin 내성균을 대상으로 mcr-1~mcr-10 의 유전자를 확인하기 위하여 mcr-1~mcr-5, mcr-6~mcr-10 2가지의 multiplex- PCR을 수행하였다. mcr1, 2는 질병관리청(Korea Disease Control and Prevention Agency, KDCA) (http://www.kdca.go.kr)에서 제시하는 특이 primer를, mcr-3~mcr-5 와 mcr-6~mcr-9은 각 Rebelo et al., Maria Borowiak et al. 의 특이 primer를 mcr-10은 Jin Seok Kim et al. 의 특이 primer를 사용 하여 PCR을 수행하였다 (12, 15, 20). mcr-1~mcr-5 multiplex-PCR 반응 조건은 94℃, 15 min; [94℃, 30 sec; 58℃, 90 sec; 72℃, 60 sec] 25 cycles; 72℃, 10 min 으로 하였고, mcr-6~mcr-10 multiplex-PCR 반응 조건은 95℃, 3 min; [95℃, 30 sec; 55℃, 30 sec; 72℃, 60 sec] 30 cycles; 72℃, 10 min 으로 하였다 (Table 1).
Table 1.
Primer pairs used to amplification of mcr genes
| Target genes | Sequences (5`→3`) | Product size (bp) | Reference |
|---|---|---|---|
| mcr-1 | F CGG TCA GTC CGT TTG TTC | 309 | http://www.kdca.go.kr |
| R CTT GGT CGG TCT GTA GGG | |||
| mcr-2 | F CTT ATG GCA CGG TCT ATG A | 650 | http://www.kdca.go.kr |
| R CAC ATT TTC TTG GTA TTT GG | |||
| mcr-3 | F AAA TAA AAA TTG TTC CGC TTA TG | 929 | Reblo AR et al. (15) |
| R AAT GGA GAT CCC CGT TTTT | |||
| mcr-4 | F TCA CTT TCA TCA CTG CGT TG | 1,116 | Reblo AR et al. (15) |
| R TTG GTC CAT GAC TAC CAA TG | |||
| mcr-5 | F ATG CGG TTG TCT GCA TTT ATC | 1,644 | Reblo AR et al. (15) |
| R TCA TTG TGG TTG TCC TTT TCT G | |||
| mcr-6 | F AGC TAT GTC AAT CCC GTG AT | 252 | Borowiak M et al. (12) |
| R ATT GGC TAG GTT GTC AAT | |||
| mcr-7 | F GCC CTT CTT TTC GTT GTT | 551 | Borowiak M et al. (12) |
| R GGT TGG TCT CTT TCT CGT | |||
| mcr-8 | F TCA ACA ATT CTA CAA AGC GTG | 856 | Borowiak M et al. (12) |
| R AAT GCT GCG CGA ATG AAG | |||
| mcr-9 | F TTC CCT TTG TTC TGG TTG | 1,011 | Borowiak M et al. (12) |
| R GCA GGT AAT AAG TCG GTC | |||
| mcr-10 | F TGC CCG TAC TTT TCA GGA TGA | 1,594 | Kim JS et al. (20) |
| R TCG CTT TCC GGA AGA TAG GC |
증폭 산물의 염기서열 분석은 carbapenemase 유전자는 Genotech (Daejeon, Korea)에서, mcr유전자는 Cosmogenetech (Seoul, Korea)에서 수행하였고, NCBI (National Center for Biotechnology informaiton)의 BLAST program을 사용하여 GenBank (http://www.ncbi.nlm.nih.gov/genebank/)의 서열과 비교하였다.
PFGE분석
PFGE 실험은 질병관리청(KDCA)에서 제공하는 표준시험법(https://pulsenetinternaitonal.org/)에 준해서 실시하였다. 분리된 균주를 NA배지(Merck, USA)에 37℃, 18‒24시간 동안 배양한 후 plug를 제작하였으며, 세척 후 plug를 1 mm 두께로 잘라 Xba I 제한효소(40 U/㎕, Roche, USA)를 사용하여 37℃에서 4시간 동안 반응시켰다. 제한효소를 처리한 plug gel을 전기영동장치(CHEF MAPPER XA Chiller, Bio-Rad, USA)를 사용해 initial time 2.16 sec, final time 63.8 sec, 전압 6 V/Cm, 120°의 조건으로 18시간 동안 전기영동 하였다. Salmonella enterica serotype Braenderup H9812 (ATCC #BAA-664)를 size marker로 사용하였으며, 실험은 분리 균주와 동일하게 실시한 후 결과 분석은 Bionumerics ver 5.1을 사용 하였다.
RESULTS
Carbapenem계 항생제 내성 장내세균속균종의 colistin내성 현황 및 특성 분석
2019~2021년 강원도의 42개 의료기관에서 Carbapenem계 항생제 내성 장내세균속균종 666주를 수집하였다. 균종 별 수집 건수는 Klebsiella pneumoniae (n=461), Escherichia coli (n=98), Enterobacter cloacae complex (n=39), Klebsiella aerogenes (n=19), Klebsiella oxytoca (n=16), Citrobacter koseri (n=15), Citrobacter freundii (n=11), Citrobacter sakazakii (n=2), 기타균종 (n=5) 이었다. 이 중 총 66주가(9.9%, colistin MIC 4~128 ㎍/㎖) colistin과 Carbapenem계 항생제에 동시 내성인 장내세균속균종(colistin-and carbapenem-resistant Enterobacteriaceae, CO-CRE)임을 확인하였다. 균종 별 colistin 내성 균주 수는 Klebsiella pneumoniae 461 주 중 45주(9.8%)가 내성 이었고, Enterobacter cloacae complex 39주 중 7주(17.9%), Klebsiella oxytoca 16주 중 7주(43.8%)가 내성이었다. 그 외에 Citrobacter koseri 15주 중 4주(26.7%), Escherichia coli는 98주 중 2주(2.0%), Citrobacter freundii 11주 중 1주(9.1%)에서 내성이 확인 되었다.
조사대상 666주 중 colistin 내성 관련 유전자인 mcr은 총 22주에서 검출되어 3.3% 의 검출률을 보였다. mcr 유전형별로는 9형이 18주로 가장 많이 검출 되었으며, 10형 3주, 8형이 1주 순이었다. 균종 별 검출 건수는 Enterobacter cloacae complex가 39주 중 14주(35.9%), Citrobacter freundii 11주 중 3주(27.3%), Klebsiella pneumoniae 461주 중 2주(0.4%) Citrobacter sakazakii 2주 중 2주(100.0%), Klebsiella oxytoca 16주 중 1주(6.3%) 순으로 검출되었다.
CO-CRE 66주 중 mcr 유전자 검출 여부를 확인한 결과 mcr-9이 5주, mcr-8, mcr-10 각 1주로 총 7주에서 검출되어 10.6% 의 검출률을 나타냈다. 균종별 mcr 검출 건수는 Klebsiella pneumoniae 45주 중 1주 Enterobacter cloacae 7주 중 6주 이었다 (Table 2).
Table 2.
Prevalence of mcr genes and colistin-resistant strains among carbapenem-resistant Enterobacteriaceae strains, Gangwon, 2019-2021
| Species | No. of isolates investigated | Prevalence of colistin-resistant isolates (%) | Prevalence of mcr genes (%) | MICa (㎎/L) | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ≤ 0.25 ~ 1 (S) | 2 (I) | ≥ 4 ~ 128 (R) | |||||||||||
| Total (S) | mcr-9 | mcr-10 | NDb | ND | Total (R) | mcr-8 | mcr-9 | mcr-10 | ND | ||||
| Klebsiella pneumonia | 461 | 9.8 | 0.4 | 327 | 1 | 326 | 89 | 45 | 1 | 44 | |||
| Escherichia coli | 98 | 2.0 | 0.0 | 94 | 94 | 2 | 2 | 2 | |||||
| Enterobacter cloacae complex | 39 | 17.9 | 35.9 | 29 | 7 | 1 | 21 | 3 | 7 | 5 | 1 | 1 | |
| Klebsiella aerogenes | 19 | 19 | 19 | ||||||||||
| Klebsiella oxytoca | 16 | 43.8 | 6.3 | 8 | 1 | 7 | 1 | 7 | 7 | ||||
| Citrobacter koseri | 15 | 26.7 | 0.0 | 11 | 11 | 4 | 4 | ||||||
| Citrobacter freundii | 11 | 9.1 | 27.3 | 10 | 3 | 7 | 1 | 1 | |||||
| Citrobacter sakazakii | 2 | 100.0 | 2 | 2 | |||||||||
| Others | 5 | 5 | 5 | ||||||||||
| Total | 666 | 9.9 | 3.3 | 505 | 13 | 2 | 490 | 95 | 66 | 1 | 5 | 1 | 59 |
CO-CRE 66주에 대한 다제내성 특성을 확인하기 위하여 총 13계열 25종의 항생제에 대한 내성률을 확인하였다. Penicillins계열 [ampicillin (AM), piperacillin (PIP)], β-lactamase inhibitors계열 [amoxicillin-clavulanic acid (AMC), amoxicillin-sulbactam (ASM)], Quinolones 계열 [nalidixic acid (NAL)]의 항생제에서 90% 이상의 내성이 확인되었다. 그리고 Cephems계열 [cefazolin (CZ), cefoxitin (FOX), cefotaxime (CTX), ceftazidime (CZD), cefepime (CEP)], Monobactams계열 [aztreonam (AZT)], Fluoroquinolones 계열 [ciprofloxacin (CIP)] 항생제에서 80% 이상의 내성이 확인되었다. colistin 외에 CRE 감염증 치료제로 사용되고 있는 Aminoglycosides계열 [amikacin (AN), gentamicin (GM), tobramycin (TOB)]의 항생제와 tigecycline (TGC)도 10% 이상의 내성이 확인 되었다 (Fig. 1).
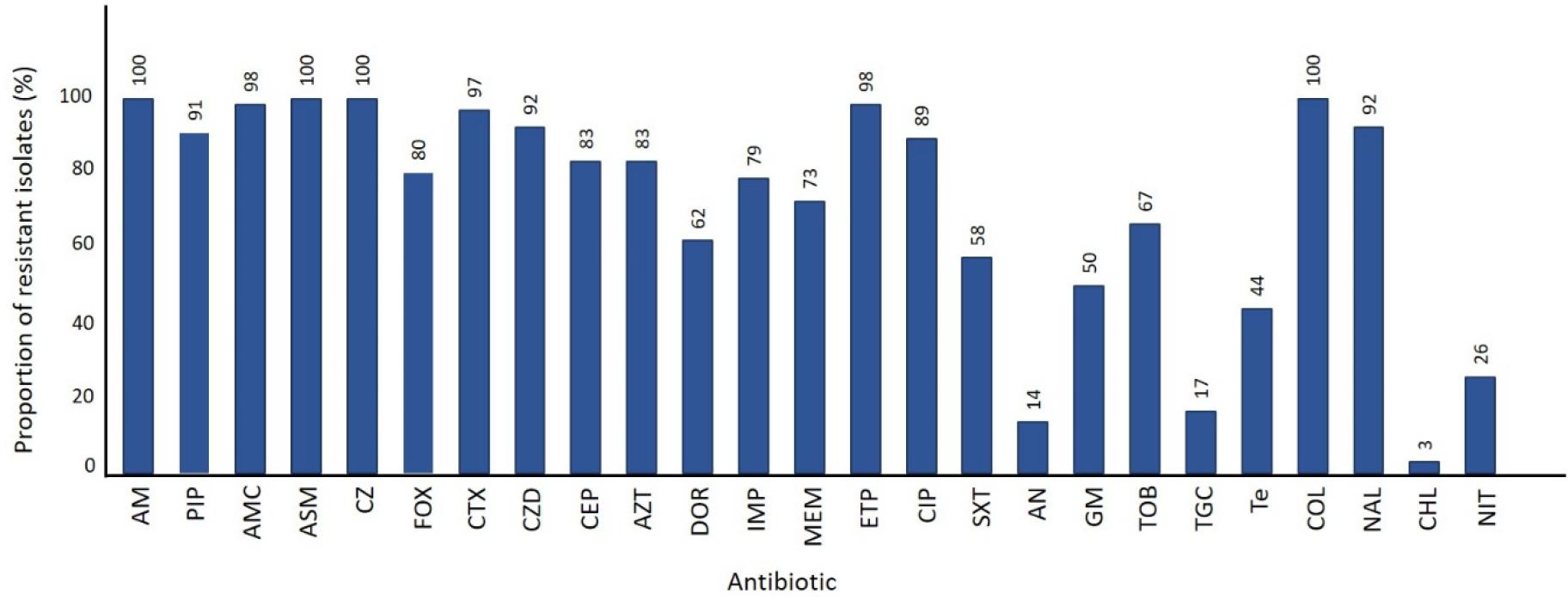
Fig. 1
Antimicrobial resistant among isolates co-resistant to carbapenem and colistin in Gangwon, South Korea (n=66).
Antibiotic susceptibility was evaluated using the broth microdilution method for doripenem, imipenem, meropenem, ertapenem, and colistin, and the disk diffusion method for other antibiotics.Abbreviations: AM, ampicillin; PIP, piperacillin; AMC, amoxicillin-clavulanic acid; ASM, amoxicillin-sulbactam; CZ, cefazolin; FOX, cefoxitin; CTX, cefotaxime; CZD, ceftazidime; CEP, cefepime; AZT, aztreonam; DOR, doripenem; IMP, imipenem; MEM, meropenem; ETP, ertapenem; CIP, ciprofloxacin; SXT, trimethoprime-sulfamethoxazole; AN, amikacin; GM, gentamicin; TOB, tobramycin; TGC, tigecycline; Te, tetracycline; COL, colistin; NAL, nalidixic acid; CHL, chloramphenicol; NIT, nitrofurantoin.
colistin과 Carbapenem계 항생제 동시 내성 장내세균속균종(Co-CRE) 간의 유전적 연관성 분석
colistin 내성 CRE균주의 전파 사례를 조사하기 위하여 pulsotype 분석을 수행하였다. 균종 별로는 Enterobacter cloacae complex 중 5주의(GWCRE19-162, GWCRE20-9, GWCRE20-27, GWCRE20-89, GWCRE20-101) pulsotype이 100% 동일하여 같은 균(Cluster 1)에 의한 전파로 추정되었다. 5주는 모두 종합병원인 A-3 기관에서 분리된 균주 였으며, 분리 시점은 1주는 2019년(7월), 4주는 2020년(1월, 2주/5월, 2주) 이었다. 또한 carbapenemase 유전자인 IMP-1과 colistin 내성 유전자인 mcr-9을 동시에 가지고 있었다 (Fig. 2A).
Klebsiella oxytoca는 pulsotype 100% 동일한 Cluster는 없었지만, 90% 이상 동일한 3주가 [(GWCRE20-18, GWCRE20-54, GWCRE20-120 (Cluster 2)] 확인되었다. 3주는 모두 종합병원인 A-3 기관에서 분리된 균주 였으며, 각각 2020년 1월, 2월, 6월에 각 1주씩 분리되었다. 또한 carbapenemase 유전자인 KPC-2를 가지고 있었으며, colistin 내성 유전자인 mcr은 확인되지 않았다 (Fig. 2B).
CRE 감염증의 주요 원인 균종인 Klebsiella pneumoniae는 4건의 전파 사례가 확인 되었다. Cluster 3은 각각 A-1, A-2, A-3 의 서로 다른 의료기관으로 부터 분리된 균주에 의한 사례이며 3주(GWCRE19-91, GWCRE19-89, GWCRE19-240)는 각 2019년 3월(2주), 10월(1주)에 서로 다른 환자 에서 분리되었다. Cluster 4도 Cluster 3과 마찬가지로 A-1, B-13의 서로 다른 의료기관에서 분리된 균주에 의한 사례이고 2주(GWCRE21-124, GWCRE21-189)는 각각 2021년 8월, 같은 해 11월에 동일한 환자로부터 분리되었다. Cluster 5는 A-1의 동일 의료기관에서 분리된 균주에 의한 사례이며 2주(GWCRE19-35, GWCRE19-66)는 각 2019년 1월과 2월에 분리되었다. Cluster 6은 A-3의 동일 의료기관에서 분리된 균주에 의한 사례이며 4주(GWCRE19-270, GWCRE19-278, GWCRE20-33, GWCRE20-34)는 각각 2019년 11월(2주), 2020년 1월(2주)에 분리되었다. Cluster 3, 4, 5의 균주들은 carbapenemase 유전자인 KPC-2를 Cluster 6은 NDM-1을 포함하고 있었으나 4건의 사례(Cluster 3~6)에서 colistin 내성 유전자인 mcr은 확인되지 않았다. 이외에 2주 이상의 유사한 pulsotype에 의한 전파 사례는 아니지만 A-3의료기관에서 분리된 Co-CRE 균주 중 GWCRE20-187은 NDM-5와 mcr-8을 동시에 포함하고 있었다 (Fig. 2C).
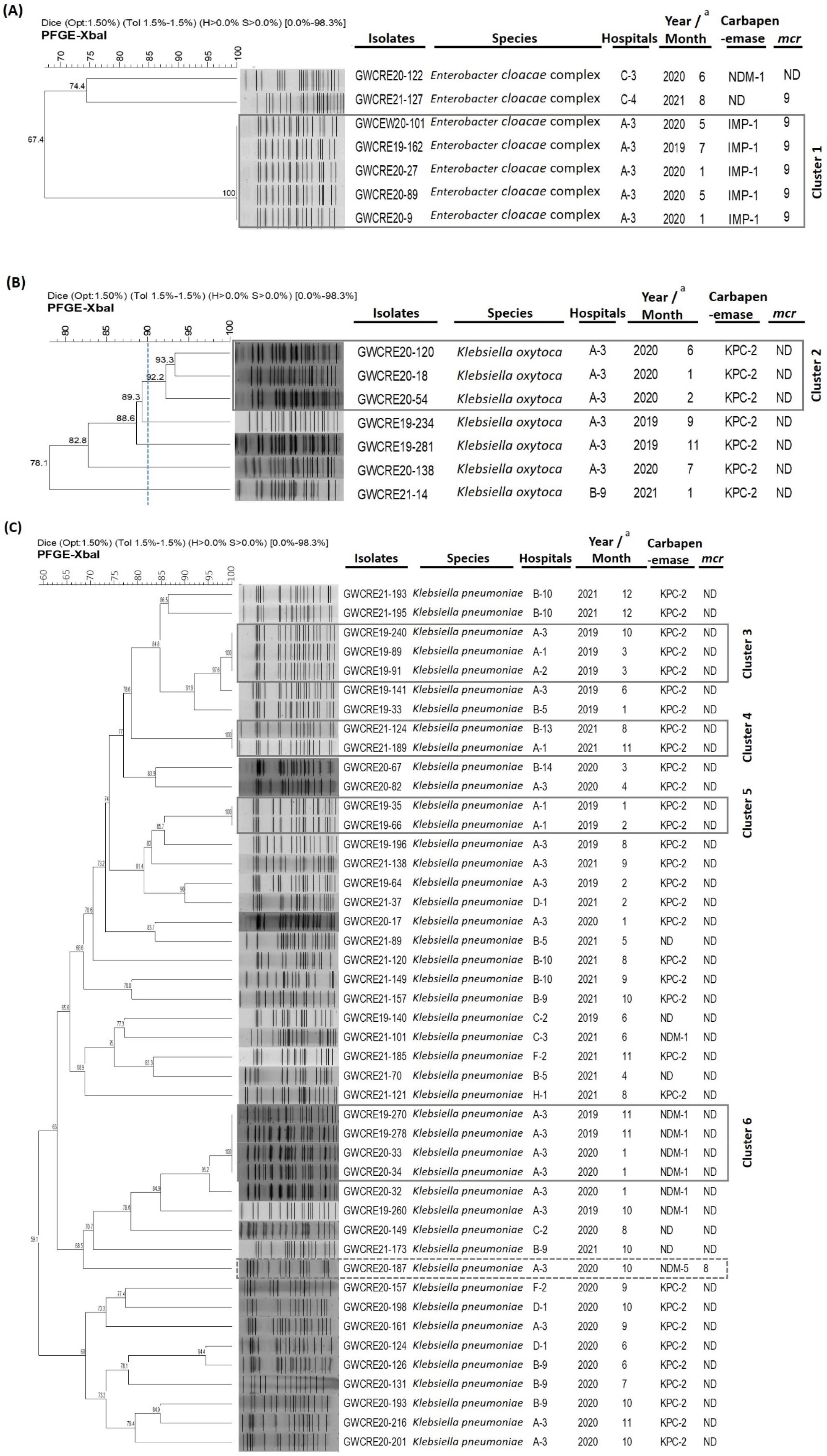
Fig. 2
Genetic relatedness and characterization of Enterobacteriaceae co-resistant to carbapenem and colistin in Gangwon, South Korea.
Dendrogram of XbaI-pulsed-field gel electrophoresis patterns (PFGE) of (A) Enterobacter cloacae complex, (B) Klebsiella oxytoca, (C) Klebsiella pneumoniae were generated using BioNumerics based on similarity analysis using the Dice coefficient and unweighted pair group method. The corresponding information, including strain ID, hospitals, isolated year/month, antimicrobial- resistance genes, are shown on the right; the square line in Fig. 2A, 2C and Fig. 2B indicated respectively the 100% and 90% similarity cut-off value for cluster analysis; the dotted square in Fig. 2C indicates isolates that have both mcr and NDM.Abbreviations: NDM, New Delhi metallo-ß-lactamase; IMP, Imipenemase; KPC, Klebsiella pneumoniae carbapenemase; mcr, Mobile Colistin Resistance; ND, mcr gene not detected. Year/Montha: Time at which samples were collected.
DISCUSSION
2019~2021년 강원도내 CRE 666주를 대상으로 colistin 내성 및 내성 유전자인 mcr에 대한 검출 현황과 내성 균주의 전파 사례 및 그 특성을 조사하였다.
colistin 내성 현황은 2019년 5.9% (16/271, 내성균주/조사대상균주), 2020년 12.8% (27/211), 2021년 12.5% (23/184) 내성으로 3년간 총 666주 중 66주가 내성으로 확인 되어 9.9%의 내성률을 확인하였다. 특히 CRE 감염증의 주요 원인 균종인 Enterobacter cloacae complex가 17.9% (7/39)를 차지하였는데, 일본에서 보고된(36주 중 50% colistin 내성) 사례보다 낮은 내성률을 보였다 (21).
mcr 검출 현황은 조사 대상 666주 중 22주에서 검출되어 3.3%의 검출률을 보였다. mcr 이 검출된 주요 균종은 Enterobacter cloacae complex로 35.9% (14/39)의 검출률을 보였다. 이는 중국의 2014~19년 CRE 분리주 1,868건에 대한 mcr 유전자 검출률을 조사한 보고에서 colistin을 임상에 적용하기 시작한 2017년을 기준으로 사용 이전은 0.4%에서 이후 1.4%로 colistin 내성이 증가한 것 보다도 1.9%p 높은 수준이었다 (22). 또한 서울시보건환경연구원의 2018~19년 서울 CRE 분리주 3,675건에 대한 연구에서 mcr 검출률은 2.2% 로 본 연구 결과가 1.1%p 높은 수준이었고, 균종별 mcr 검출률 비교에서 Enterobacter cloacae complex 가 가장 높은 검출률(20.5%)을 보인 것은 본 연구결과와 일치하였다 (20). mcr 유전자는 균주 간 수평 전파가 가능하기 때문에 mcr 유전자를 보유한 균주가 colistin에 내성이 없더라도 다른 균주로 옮겨간 이후 새로운 균주의 유전적 환경에 따라 내성 표현형을 나타낼 수 있는 가능성이 있으므로 전파 차단을 위한 지속적인 감시가 필요하다. 특히, CO-CRE 66주 중 7주에서 mcr-8 (1주), mcr-9 (5주), mcr-10 (1주)이 검출되었는데, 이는 내성만 있는 균주 또는 내성유전자인 mcr 만 있는 균주 보다 두가지 조건을 모두 만족하는 경우이므로 내성률 증가를 보다 가속화 할 수 있을 것으로 판단된다.
mcr 유전자가 검출되지 않은 colistin 내성균주들의 경우 mcr 이외의 다른 내성 기작을 통하여 내성을 획득했을 것으로 생각된다. colistin 내성 획득은 mcr 이외에도 다양한 유전자 와 오페론들이 관여하는데, 세균 세포벽의 lipopolysaccharide (LPS) 변이를 유발하는 pmrC 와 pmrE 유전자, pmrHFIJKLM 오페론, PmrAB 와 PhoPQ가 관여하는 two-component systems (TCSs), capsule 생합성을 증가시켜 내성을 획득하는데 관여하는 Cpx 와 Rcs 그리고 efflux pump KpnEF, AcrAB 등의 활성화로 인하여 colistin 내성이 증가하는 것으로 보고 되어있다 (23).
Co-CRE로 확인된 66주에 대한 13계열 25종에 대한 항생제의 다제내성 특성을 조사한 결과 10~23제의 항생제에 내성을 나타냈으며, Penicillins계열과 β-lactamase inhibitors, Quinolones 계열의 항생제에서는 90% 이상의 내성이 확인되었다. 66주 모두 3계열 이상의 항생제에 내성을 나타내어 다제내성 특성이 있음을 확인하였으며, colistin과 함께 CRE치료제로 사용되고있는 Aminoglycosides계열의 항생제에서도 10% 이상의 내성이 확인되어 내성 양상에 대한 지속적인 관심이 요구된다.
본 연구에서 주목할만한 점은 Co-CRE의 강원도내 의료기관 4곳에서 원내 또는 지역사회 집단 전파 의심 사례를 확인한 것 이다. 동일한 의료기관내(A-1, A-3) 전파 4사례 [Enterobacter cloacae complex (cluster 1), Klebsiella oxytoca (cluster 2), Klebsiella pneumoniae (cluster 5, 6)] 의료기관 간의(A-1, A-2, A-3, B-13) 전파 2사례 [Klebsiella pneumoniae (Cluster 3, 4)] 이었다. 본 연구팀은 cluster 1에 포함되는 Enterobacter cloacae complex [Enterobacter asburiae (GWCRE20-27, GWCRE20-89, GWCRE20-101)]인 Co-CRE 균주의 동일 의료기관내 전파 사례를 이미 보고한 바 있으며, 이번 연구에서 pulsotype이 동일한 2주 (GWCRE19-162, GWCRE20-9)를 추가로 확인 하였다 (4). 프랑스, 인도와 브라질에서도 colistin과 Carbapenem계 항생제에 동시 내성인 Klebsiella pneumoniae에 의한 의료기관내 전파 사례가 보고되었다. 프랑스와 인도 사례에서는 OXA-48을 가지는 균주에 의한 집단 발생이었고, 브라질 사례는 본 연구와 마찬가지로 KPC를 가지는 균주에 의한 것 이었다 (3, 5, 6).
총 6건의 전파사례에서 확인된 균주 모두 carbapenemase 유전자인 IMP-1 (Cluster 1), KPC-2 (Cluster 2~5) 또는 NDM-1 (Cluster 6)을 포함 하고 있었지만, Enterobacter cloacae complex (Cluster 1)에 의한 사례를 제외한 Klebsiella oxytoca (Cluster 2), Klebsiella pneumonia (Cluster 3~6)에 의한 사례에서 mcr은 검출되지 않았다. 따라서 이러한 균주 들의 colistin 내성 발현과 관련된 기작에 관한 연구가 추가적으로 수행 되어야 한다.
이번 연구의 한계점은 환자들의 colistin 처방 여부 및 복용 기간, 임상증상등의 보다 자세한 임상 정보와, 의료기관내 환자들 간의 역학적 연관성에 관한 정보가 매우 제한적이라는 점이다.
하지만 본 연구를 통하여 CRE 감염증의 치료용 항생제인 colistin에 대한 내성 균주를 확인하였으며, 일부 균주는 내성유전자인 mcr을 가지고 있었다. 그리고 이들 내성 균주의 의료기관 또는 지역사회 집단 전파 사례가 확인되어, 추가적인 전파 차단을 위한 의료기관 내 감염관리 기준 강화가 필요한 상황임을 알 수 있었다. 또한 colistin의 내성 증가를 방지하기 위한 대체 항생 요법 및 치료제의 개발이 시급하며, 내성발현기작에 관한 분자유전학적 연구와 지속적인 내성 감시가 요구된다.




