INTRODUCTION
엔테로바이러스는 Picornaviridae과 Enterovirus속 정이십면체 형태의 외막이 없는 약 7.4 kb의 작은 양성 단일 가닥 RNA 바이러스이다. 엔테로바이러스는 세포배양을 통하여 바이러스를 분리하고 중화항체법에 의한 혈청형 동정법을 이용하여 약 70가지 이상의 혈청형들이 밝혀져 있고, 실험 동물의 조직 배양과 질병 양상에 따라 폴리오바이러스(Polioviruses, PV) 및 콕사키바이러스 A (Coxsakievirus A, CV-As)와 콕사키바이러스 B (Coxsakievirus B, CV-Bs), 에코바이러스(Echoviruses, ECV), 기타 엔테로바이러스(Human Enteroviruses)로 분류되고 있다 (1). 최근에는 RNA dependent RNA-polymerase 특정 부위의 염기서열을 결정하고, 계통분석에 의한 염기서열 간의 유전적 상동도를 기반으로 폴리오바이러스(3종)와 엔테로바이러스 A (Human Enterovirus A, HEV-A), 엔테로바이러스 B (Human Enterovirus B, HEV-B), 엔테로바이러스 C (Human Enterovirus C, HEV-C), 엔테로바이러스 D (Human Enterovirus D, HEV-D)와 라이노바이러스(Rhinovirus A ~ C)로 분류하고 있다 (2). HEV-A에는 11가지 혈청형의 CV-A와 EV-A71 등이 속하며, HEV-B는 모든 CV-Bs와 ECV, CV-A9 등이 포함된다. HEV-C는 PV-1~3과 CV-A1 등이 포함되며, HEV-D는 EV-D68, EV-D70 등이 속한다 (3, 4). 바이러스의 유전자 중 한 부분을 차지하는 virus protein 1(VP1) region은 주요 항원 부위이면서 중화결정자로서의 기능을 하며, 엔테로바이러스 동정에 유전적 상관관계가 있어 genotyping에 이상적인 target 부분으로 사용된다 (5).
엔테로바이러스는 전세계적으로 퍼져있고 주로 분변-구강의 경로를 통하여 사람에게 전파되며 소화기를 통해 감염된 후 인후두 부위나 소장의 림프절에서 일차적으로 증식하고 신체의 각 장기로 이동하는 것으로 알려져 있다 (6). 임상증상은 무증상 감염에서부터 발열, 피부발진, 인후염, 수족구병, 헤르페스목구멍염, 심근염 또는 무균성 수막염과 뇌염을 일으킬 정도로 매우 다양한 증상을 보인다고 알려져 있으며 (7), 특히 엔테로바이러스 71의 경우 신경계 합병증을 유발하며 폐수종이나 뇌질환 합병증으로 사망하는 경우도 보고되고 있다 (8, 9). 국내에서는 2009년 엔테로바이러스 71에 의한 사망자 발생으로 엔테로바이러스에 대한 관심이 시작되었고 (10) 2014년부터 2016년에는 엔테로바이러스 D68에 의한 폐렴이 미국에서 유행하였으며 (11), 일본, 중국 및 홍콩 등 아시아에서도 매년 엔테로바이러스 감염증이 꾸준히 발병하는 등 전세계적으로 엔테로바이러스 감염증의 유행이 보고되고 있다 (12, 13). 엔테로바이러스 감염은 주로 여름철에 호발하고 대부분 10세 이하의 영유아에서 주로 검출되는 특성이 있고 유행하는 혈청형은 지역과 시기에 따라 다르다 (14). 우리나라에서는 질병관리본부에 의뢰된 엔테로바이러스 검체를 대상으로 시행한 연구에서 엔테로바이러스 양성율이 1999년 41%, 2003년 31%, 2005년 43%, 2008년 56%로 약 3년 주기의 유행이 보고된 바 있으며 주기적인 유행으로 인한 사회, 경제적으로 손실을 유발하여 엔테로바이러스에 대한 연구의 중요성이 대두되고 있다 (15).
본 연구의 목적은 최근 부산지역에서 검출된 엔테로바이러스를 대상으로 분자생물학적인 분석을 통하여 부산지역에서 유행하고 있는 엔테로바이러스의 유전적 다양성을 확인하고, 염기서열 데이터베이스 구축을 위한 기초 자료로써 제공하고자 하였으며 이를 통하여 감염병 예방 대책 수립과 새로운 진단법 개발 및 백신개발 등에 대한 자료로 제공하고자 한다.
MATERIALS AND METHODS
연구 대상
2017년부터 2022년까지 부산시내 3개 병원 소아과에 내원한 엔테로바이러스 감염 의심환자의 대변, 뇌척수액, 인후도찰물을 stool box, cryovial, virus transport medium에 각각 담아 4℃를 유지하여 운송하였다. 총 1,412건에 대하여 엔테로바이러스 유전자 검사를 실시하였고 유전자가 검출된 검체를 대상으로 염기서열 분석을 수행하였다.
검체 전처리 및 바이러스 RNA 추출
뇌척수액 및 인후도찰물의 경우 전처리 없이 그대로 검체로 사용하였으며, 대변의 경우 glass beads (8개/1 g, 직경3 ㎜)를 넣은 centrifuge tube에 pH 7.4의 phosphate buffered saline (PBS, Sigma, USA) 완충용액을 만들어 대변 2 g 넣고 20분 동안 강하게 진탕하였다. 그 후 3,000 rpm (1248R, Labogene, Korea), 4℃에서 20분간 원심분리하고 상층액을 검체로 사용하였다. 검체 140 ㎕을 QIAamp Viral RNA mini Kit (Qiagen, Germany)를 이용하여 제조사의 매뉴얼에 따라 RNA를 추출하여 cDNA합성을 위한 template로 사용하였다.
엔테로바이러스 유전자 검출
5′-noncoding region의 실시간 RT-PCR
엔테로바이러스의 5′non-coding region을 표적으로 하여 real-time RT-PCR을 수행하였고 증폭산물의 Ct (threshold cycle)값이 35 이하이면 양성으로 판정하였으며, 검체로부터 엔테로바이러스가 검출되었음을 의미하였다. primers (EN1: 5'-CCCTGAATGCGGCTAA-3'; EN2: 5'-ATTGTCACCATAAGCAGCC-3';), probe (EN3: FAM-CCGACTACTTTGGGTGT-BHQ)를 포함한 PowerChekTM Human Enterovirus Real-time PCR Kit (Kogene biotech, Korea)를 사용하였으며 검체로부터 추출한 RNA 5 μl를 첨가하여 실험하였다. 실시간 RT-PCR 조건은 50℃에서 30분, 95℃에서 10분 1회 수행한 다음, 95℃에서 15초, 60℃에서 1분으로 40회 실시하였으며, Thermal cylcer (7500Fast, Applied Biosystems, USA)를 이용하여 증폭 산물을 실시간으로 검출하였다.
VP1 region의 nested RT-PCR
VP1 영역의 증폭을 위하여 Table 1에 제시된 primer가 포함된 LiliF EV VP1 1st RT-PCR Kit (iNtRON, Korea)를 이용하였다. 1차 RT-PCR 반응액은 각각의 tube에 DNases/RNase Free Water를 18 ㎕씩 첨가하고 양성대조, 음성대조, 추출된 RNA 2 ㎕를 각각 첨가하여 총 부피가 20 μl가 되도록 하였다. 1차 RT-PCR 조건은 Thermal cycler (ProFlex, Applied Biosystems, USA)를 이용하여 42℃에서 45분, 94℃에서 5분 동안 1회 수행, 94℃에서 30초, 55℃에서 30초, 72℃에서 40초 동안 40회 반복한 후 72℃에서 5분 동안 수행하였다. 2차 PCR 반응액은 2차 PCR kit에 1차 PCR 반응산물 1 μl와 DNase/RNase free water 19 μl를 넣어 총 부피가 20 μl가 되도록 하였다. 2차 RT-PCR (LiliF EV VP1 2nd RT-PCR Kit (iNtRON, Korea)) 조건은 94℃에서 5분동안 1회 수행, 94℃에서 30초, 55℃에서 30초, 72℃에서 40초 동안 40회 반복한 후 72℃에서 5분 동안 수행하였다. 증폭된 PCR 산물들은 전자동 전기영동장치(QIAxcel Advanced, Germany)에서 350~371 bp 밴드를 확인하였다. 검체 중 PCR 증폭이 확인된 산물은 전문 업체(Macrogen, Daejeon, Korea)에 의뢰하여, 염기서열 분석(Sanger Sequencing)을 진행하였다.
Table 1.
Primers information for RT-PCR and sequencing of human enterovirus detection
엔테로바이러스 유전자형 확인 및 상동도 분석
증폭된 PCR 산물들은 정제시킨 후 염기서열을 결정하였으며, 각각의 VP1 유전자에 대한 염기서열은 NCBI의 BLAST search program (Basic Local Alignment Search Tool, BLAST: Basic Local Alignment Search Tool (http://blast.ncbi.nlm.nih.gov/Blast.cgi))을 이용하여 기존에 보고된 바이러스 염기서열들과 상동도를 확인하였으며, 확인된 염기서열 99건은 Genbank (http://www.ncbi.nim.nih.gov/)에 등록된 엔테로바이러스 분리주들과 비교하여 국가와 시기별로 분류하였고 겹치는 염기서열들 중에서는 결정시기가 빠른 것으로 추출하였다 (Table 2, 3). 염기서열들은 MEGA (Molecular Evolutionary Genetics Analysis version 6.0) software를 이용하여 Clustal-W(1.6) method로 서열 정렬 후 Neighbor-Joining Method에 의해 염기서열간의 유전적 거리를 추론하여 phylogenetic tree를 작성하였으며, bootstrap 1,000으로 설정하여 수행하였다.
Table 2.
Reference enterovirus A strains from genbank used in this phylogenetic analysis
Table 3.
Reference enterovirus B strains from genbank used in this phylogenetic analysis
RESULTS
엔테로바이러스 유전자 확인
부산지역에서 2017년부터 2022년까지 임상적으로 엔테로바이러스 감염증으로 의심되는 환자의 대변 602건, 인후도찰물 484건, 뇌척수액 326건, 총 1,412건의 검체에서 RNA를 추출하여 5′-noncoding region의 실시간 RT-PCR을 수행하여 128건의 검체에서 증폭을 확인하였고, VP1 region RT-PCR을 수행하여 99건의 검체에서 증폭산물인 350-371bp band를 확인하였다. 총 99건의 검출 중 대변에서 66건이(검출률 11.0%), 인후도찰물이 20건이(검출률 4.1%), 뇌척수액 13건(검출률 4.0%)이 검출되었으며 대변에서의 검출률이 가장 높았다.
엔테로바이러스 유행양상 분석
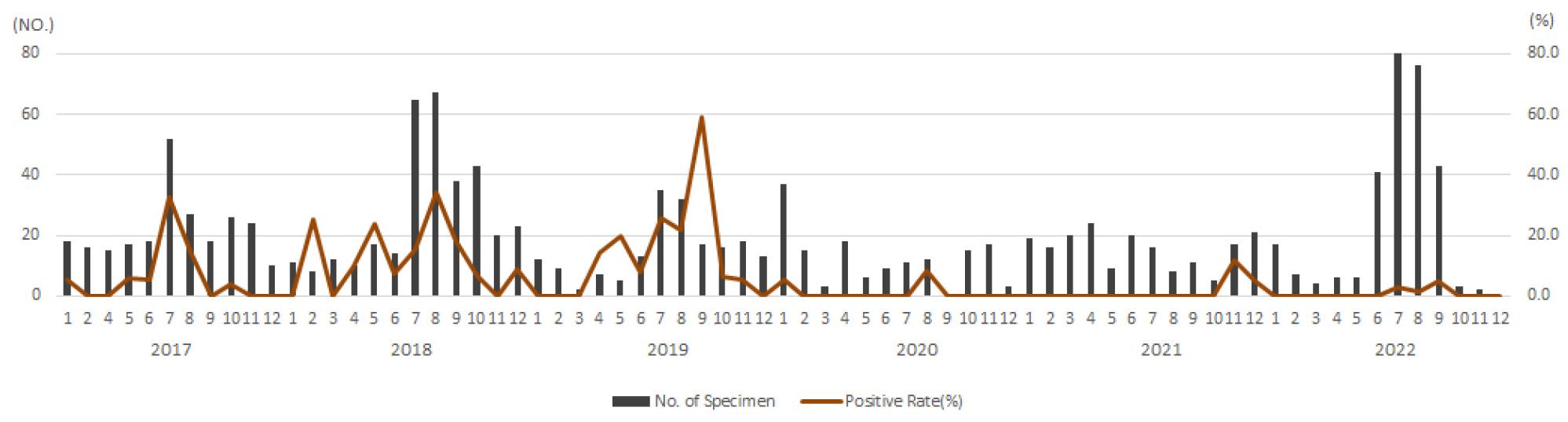
총 1,412건의 검체 중 128건(9.1%)에서 엔테로바이러스가 검출되었다. 연도별로는 2017년에는 엔테로바이러스 감염의심 환자 검체 259건에서 25건(9.7%)에서 엔테로바이러스가 검출되었고, 2018년의 경우 328건 중 60건(18.3%), 2019년 179건 중 31건(17.3%)이 검출되었다. 2017년부터 2019년까지 부산지역에서 엔테로바이러스가 매년 유행하고 있음을 알 수 있었다 (Table 4).
Table 4.
Genotypes of human enterovirus detected from Busan, during 2017~2022
본 연구에 사용된 검체 1,412건 중 752건이 6월부터 9월까지 4개월간 의뢰되어 전체건수의 53.3%를 차지하였다. 엔테로바이러스는 5월부터 검출률이 증가하기 시작해서 7월(14.6%), 8월(16.7%), 9월(15.0%)에 높게 나타나 엔테로바이러스의 검출이 하절기에 집중되어 나타난 것을 확인하였다 (Fig. 1). 2007년부터 2009년까지 부산지역에서 검출된 엔테로바이러스의 분석 결과를 보면 2007년엔 8.6%의 검출률을, 2008년엔 8.7%의 검출률을, 2009년에는 9.0%의 검출률을 보이고 있어 매년 엔테로바이러스가 유행하였음을 확인할 수 있었고, 유행시기 또한 6월 (15.5%), 7월 (15.8%), 8월 (15.3%)로 하절기에 집중된 것을 알 수 있었다 (16). 충남지역 엔테로바이러스 연구결과에서도 2005년부터 2006년까지 매년 엔테로바이러스가 유행하였음을 확인할 수 있었고, 유행 시기 또한 하절기였음을 알 수 있었다 (17). 인천지역에서 수행한 연구에서도 앞선 연구들과 마찬가지로 엔테로바이러스의 유행양상이 유사한 것을 확인할 수 있었다 (18). 이것으로 볼 때 지역적으로 약간의 차이는 있으나, 지역마다 매년 엔테로바이러스가 하절기에 유행하고 있음을 알 수 있었다.
엔테로바이러스 유전자형 결정 및 계통분석
엔테로바이러스의 5’-noncoding region에 대한 실시간 RT-PCR 결과 양성으로 확인된 128건에 대한 RT-PCR로 VP1 유전자를 증폭한 후 염기서열을 분석하여 혈청형 및 유전자형이 결정된 것은 99건이었고 29건은 Untyped였다. 검출된 유전자형 99건 중 93건은 엔테로바이러스 유전자형으로 분류되었고, 나머지 6건은 라이노바이러스 유전자형으로 분류되었다. 엔테로바이러스 유전자형으로 확인된 93건은 HEV-A, HEV-B, HEV-D 세 종류였으며 HEV-A로 분류된 혈청형은 7종(CV-A2, CV-A4, CV-A5, CV-A6, CV-A10, CV-A16, EV-A71) 총 30건이었으며, HEV-B로 분류된 혈청형은 10종(CV-A9, CV-B2, CV-B3, CV-B4, CV-B5, E-9, E-11, E-13 E-25, E-30) 총 63건, HEV-D로 분류된 혈청형은 1종(EV-D68), 1건이었고, HEV-C는 검출되지 않았으며 라이노바이러스는 총 2건이 검출되었다.
년도별로 검출된 혈청형은 Table 4와 같으며 2017년에는 CV-A2 1건, CV-A5 1건, CV-A9 2건, CV-A10 11건, CV-B2 4건, CV-B4 2건이었다. 2018년에는 CV-A4 2건, CV-A6 1건, CV-A16 1건, EV-A71 3건, CV-B3 1건, CV-B5 19건, E-11 3건, E-13 2건, E-25 2건, E-30 7건이었다. 2019년에는 CV-A2 1건, CV-A6 4건, CV-A16 1건, EV-A71 2건, E-9 3건, E-11 4건, E-30 14건이었다. 2020년에는 EV-A71 2건이었고 2022년에는 EV-D68 1건이 확인되었다. 그 외 Rhinovirus A 4건, Rhinovirus B 1건도 확인되어 해마다 출현하는 엔테로바이러스의 유전자형 및 혈청형은 다양한 것으로 확인되었다.
엔테로바이러스 분리주의 염기서열과 비교 염기서열의 상동도 분석 결과
부산 유행 Human Enterovirus A (HEV-A) 염기서열과 비교염기서열(Table 2)의 계통 분석 (Fig. 2)

Fig. 2
Phylogenetic analysis of the partial sequences of the vp-1 region of human enterovirus A from 30 isolates from Busan and 19 reference strains. Reference strains are listed by abbreviations: CV, Coxsakievirus; EV, Enterovirus. All references sequences were obtained from the Genbank. The tree was generated using the neighbor-joining methods and the trees were constructed using the MEGA, version 6.0. The bar represents 0.05 genetic distance unit.
2017년과 2019년에 검출된 CV-A2 분리주는 2015년의 홍콩, 태국의 분리주들과 상동도가 높았으며, 중국에서 분리된 염기서열과도 유사한 상동도를 나타내었다. 부산지역에서 2018년도에만 검출되었던 CV-A4 분리주는 2013년 중국의 분리주들과 상동도가 매우 높았으며, 2017년도에만 검출되었던 CV-A5 분리주는 중국 및 미국의 분리주와 높은 상동도를 보였다. 2018년과 2019년에 검출된 CV-A6 분리주는 2007년 대만의 분리주와 상동도가 높았으며 2013년 중국의 분리주들과도 높은 상도도를 보였다. 2017년도에 가장 많이 검출되었던 CV-A10 분리주 11건은 2013년과 2009년의 중국 분리주들과 상동도가 높았으며 2012년 중앙아프리카에서의 분리주와도 유사한 상동도를 보였다. 2018년과 2019년에 검출되었던 CV-A16 분리주는 2014년 중국의 분리주들과 상동도가 매우 높았으며 2011년 일본의 분리주와도 높은 상동도를 보였다. 마지막으로 2018년부터 2020년까지 검출되었던 EV-A71 분리주의 경우 2012년 캄보디아의 분리주와 높은 상동도를 보였고 중국, 태국의 분리주들과도 유사한 상동도를 보이는 것으로 확인되었다.
부산 유행 Human Enterovirus B (HEV-B) 염기서열과 비교염기서열(Table 3)의 계통 분석 (Fig. 3)
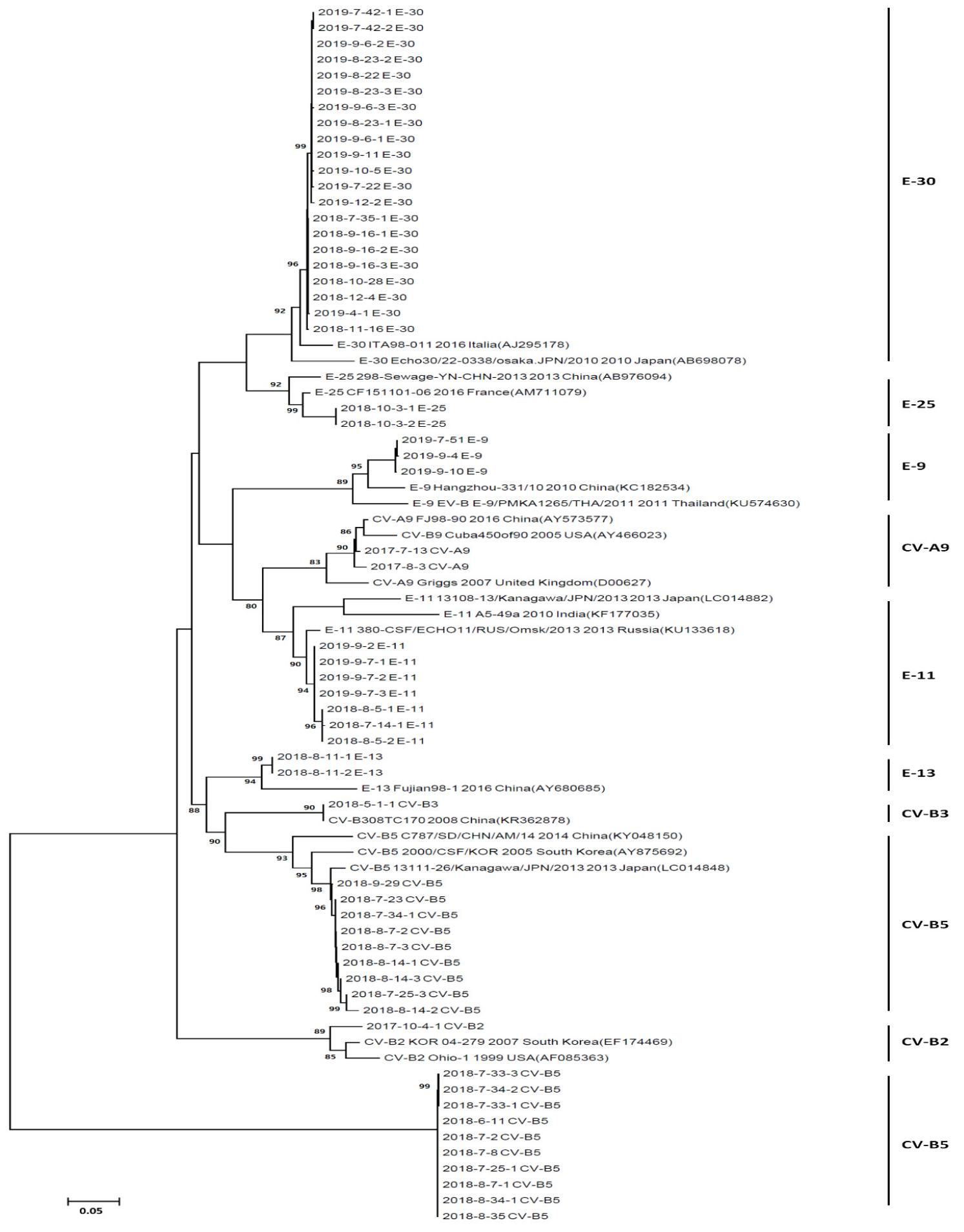
Fig. 3
Phylogenetic analysis of the partial sequences of the vp-1 region of human enterovirus B from 58 isolates from Busan and 19 reference strains. Reference strains are listed by abbreviations: CV, Coxsakievirus; E, Echovirus. All references sequences were obtained from the Genbank. The tree was generated using the neighbor-joining methods and the trees were constructed using the MEGA, version 6.0. The bar represents 0.05 genetic distance unit.
2017년에 검출된 CV-A9 분리주는 2007년의 영국의 분리주와 상동도가 높았으며, 미국, 중국의 분리주들과도 상동도가 높았다. 2017년에 검출된 CV-B2 분리주는 2007년 한국형 분리주와 상동도가 매우 높았고 미국의 분리주와도 유사한 상동도를 나타내었다. 2018년에 검출된 CV-B3 분리주는 2008년 중국의 분리주와 유사한 상동도를 나타내었다. 2018년 가장 많이 검출된 CV-B5 분리주의 경우 2013년 일본의 분리주와 상동도가 매우 높았고 2005년 한국, 2014년 중국의 분리주와도 유사한 상동도를 나타내었다. 2019년에 검출된 E-9 분리주는 2011년 태국 및 중국의 분리주와 높은 상동도를 보였다. 2018년과 2019년에 검출된 E-11 분리주는 2013년 러시아 분리주와 높은 상동도를 보였고 일본, 인도의 분리주들과도 유사한 상동도를 보였다. 2018년도 검출된 E-13 분리주는 2016년 중국의 분리주와 매우 유사한 상동도를 보였고 2018년도에 검출된 E-25 분리주의 경우 2016년 프랑스의 분리주와 높은 상동도를 보였다. 마지막으로 2018년과 2019년에 가장 많이 검출된 E-30 분리주는 2016년 이탈리아의 분리주와 높은 상동도를 보였고 2010년 일본의 분리주와도 유사한 상동도를 보이는 것으로 확인되었다.
DISCUSSION
연도별 엔테로바이러스 유행은 Fig. 1과 같으며, 2017년부터 2019년까지 부산지역에서 엔테로바이러스가 매년 유행하고 있음을 알 수 있었다. 하지만 2020년부터 2022년, 최근 3년간 엔테로바이러스 감염의심 환자 검체는 646건이었고 검출은 12건(1.9%)이었으며, 최근 3년간의 낮은 엔테로바이러스 검출률은 코로나19의 유행으로 인해 강화된 위생규정, 마스크 착용, 사회적 거리두기 실시 등의 영향으로 사료된다 (19).
Kim 등의 연구에서 보면 2007년부터 2009년까지 부산지역에서 유행한 엔테로바이러스의 유전자형은 HEV-A와 HEV-B 두 종류의 유전자형이 부산지역에서 유행하였음을 알 수 있었고, HEV-A로 분류된 혈청형은 6종(CV-A2, CV-A4, CV-A5, CV-A10, CV-A16, EV-A71) 총 31건이었으며, HEV-B로 분류된 혈청형은 10종(CV-A9, CV-B1, CV-B2, CV-B3, CV-B4, E-6, E-7, E-9, E-11, E-30) 총 41건으로 확인되어 다양한 엔테로바이러스 혈청형이 부산지역에서 유행하는 것을 알 수 있었다 (17). 최근 미국 등에서 소아에 폐렴, 급성 이환성 척수염 등을 유발한다고 보고되고 있는 엔테로바이러스 D68이 2022년 부산지역에서도 검출되는 것으로 보아 매년 유행하는 엔테로바이러스의 유전자형 및 혈청형은 다소 변화하고 있음을 알 수 있었다 (13). Park 등의 연구에서 2005년도에 충남지역 유행을 주도한 E-18 의 경우 내부적으로 95.8~99.2%의 상동도를 보였고, 동일한 바이러스 종이 같은 시기에 유행하였음에도 불구하고 4.2%의 다양성을 보였으며 (17),7CV-B3의 경우 내부적으로 80.5~90.0% 까지 다양한 상동도를 보였다 (20). 또한 2006년도에 충남지역 유행을 주도한 E-5의 경우 내부적으로는 97.7~99.4%의 상동도를 보였고, 외국의 원형주들과의 상동도 비교에서는 79.4~85.9%의 수치를 보고하였다 (17). 유전자형간의 핵산염기서열 상동도 비교를 실시한 결과 서로 같은 혈청형 간에도 상동도의 차이를 보이는 것은 엔테로바이러스가 한가닥의 RNA를 가져 돌연변이율이 매우 높다는 보고가 있다 (21).
부산지역에서 검출된 엔테로바이러스 분리주와 비교 분리주의 상동도를 비교한 결과를 토대로 그린 계통수를 보면 대부분 중국이나 동남아시아에서 검출되다가 우리나라에 도달하여 우리나라에서 검출되었다고 추정은 가능하나 정확한 원형이라고 확증할 수는 없었다. 또한 같은 지역인 부산에서 발견되었음에도 상동도에 차이를 보이는 경우가 있었는데 이것은 단일가닥을 가지고 있는 RNA 바이러스인 엔테로바이러스가 이중 가닥을 가진 DNA 바이러스보다 빠르게 돌연변이가 진행되는 특성 때문에 같은 지역에서 검출된 염기서열일지라도 상동도에 차이가 발생한다고 볼 수 있을 것이다 (22). 또한 CV-B5, E25 및 E30은 환경 검체로부터 분리된 환경 분리주가 비교 염기서열에서 확인되었고, 그 환경 분리균주가 인체에서 분리된 분리균주와 상동도를 나타내고 있는 것으로 보아 이는 엔테로바이러스가 환경 검체로부터 영향을 주고받아 인체에 감염시킬 수 있다는 최근의 연구 결과로 볼 때 (23, 24) 환경 검체에 대한 엔테로바이러스의 연구가 인체 검체에만 한정된 것이 아니라 환경 검체에 대한 감시도 필요할 것으로 사료된다.
본 연구를 통해서 최근 부산지역에 검출된 엔테로바이러스 유전자형들 사이에 다양한 상동도를 보이는 결과를 확인할 수 있었으며, 이를 볼 때 부산지역에서 유행하는 엔테로바이러스 내에 유전자 변이 발생이 가능하였음을 추측해볼 수 있어 향후 엔테로바이러스이 변이에 대한 연구를 추가해볼 것을 건의한다. 또한 엔테로바이러스의 발생양상 파악 및 유전자형 분석은 엔테로바이러스의 유행 추세와 패턴을 확인하고 검출되는 유전자형에 대한 정보를 제공함으로써 공중보건 및 임상적 관점 모두에서 유용한 수단이 될 수 있다. 본 연구를 통해 부산지역에서 유행하는 엔테로바이러스의 유전자형을 확인하고 엔테로바이러스 예방관리를 위한 공중보건학적 자료로 활용될 수 있기를 기대한다.