INTRODUCTION
홍역(Measles)은 가래, 비말과 같은 호흡기 분비물을 통한 전파 또는 공기 중의 홍역바이러스(Measles virus)를 통해 전파가 되는 전파력이 매우 높은 감염병 중 하나이다 (1, 2, 3). 홍역의 기초감염재생산지수(R0)는 12~18로 알려져 있으며, 이는 홍역에 대한 면역력이 없는 집단이 홍역에 노출될 경우 90% 이상이 홍역에 이환 될 수 있음을 의미한다.
홍역의 증상은 고열, 기침, 콧물, 결막염, 피부발진 등이 있으며, 특히 구강점막에 하얀색 반점으로 나타나는 코플릭 반점(koplik spots)이 특징적인 임상증상 중 하나이다 (4, 5). 증상의 경과에 따라 발진이 발생하기 전 발열, 코플릭 반점 등이 발생하는 전구기, 피부발진이 목 뒤에서부터 발생하여 온몸으로 퍼지는 발진기, 발진이 사라지며 회복되는 회복기 세 단계로 구분되며, 발진 발생 4일 전부터 발진 발생 4일 후까지 전파력이 강한 것으로 알려져 있다. 홍역 환자의 대부분은 증상이 사라지면서 회복이 되고 홍역에 대한 영구면역을 얻는 것으로 알려져 있지만, 면역력이 약한 환자나 영유아의 경우 홍역으로 인해 사망까지 이를 수 있는 것으로 알려져 있고, 2019년에는 전 세계적인 대유행으로 20만 명 이상이 사망한 것으로 보고되었다 (6).
홍역을 유발하는 홍역바이러스는 paramyxoviridae의 morbillivirus에 속하는 단일가닥 음성(-) RNA 바이러스로 haemagglutinin (H), fusion (F), matrix (M), nucleoprotein (N), phosphoprotein (P), large protein (L)과 같은 6개의 구조단백질로 구성되어 있다 (7, 8, 9). 바이러스의 유전적 다양성은 nucleoprotein을 암호화하는 N gene에서 가장 크고 최대 12%의 다양성을 보인다고 알려져 있다 (10, 11, 12). 홍역바이러스는 1개의 혈청형과 23개의 유전형(A, B1-B3, C1-C2, D1-D10, E, F, G1-G3, H1-H2)으로 구분되고 있지만, 2018년 전 세계를 대상으로 한 보고에 따르면 다섯 종류의 유전형(A, B3, D4, D8, H1)이 확인되었다 (13). 그 중 A 유전형은 백신주이며, 나머지 네 종류의 유전형은 홍역이 풍토병으로 남아있는 국가별로 토착화 되어있는 것으로 보고되고 있다.
1963년 홍역 백신이 개발되기 전에는 매년 약 260만 명이 홍역으로 사망하였으나, 홍역 백신의 개발 및 도입으로 전 세계적인 홍역 환자의 발생 및 사망 수가 크게 감소하였다 (14, 15). 현재 홍역 백신은 2회 접종이 권장되고 있으며 접종 완료 시 97%의 예방효과를 보이는 것으로 알려져 있다 (16). 홍역바이러스는 단일 혈청형이고 홍역 백신의 예방효과는 매우 뛰어나지만 홍역의 전파력이 매우 강하기 때문에 인구의 95% 이상이 면역을 획득해야 홍역의 전파를 막을 수 있기 때문에 예방 및 관리를 위해 많은 노력이 필요한 감염병 중 하나이다 (17, 18).
우리나라는 세계보건기구(World Health Organization, WHO)의 홍역퇴치 기준에 따라 2014년 3월 홍역퇴치국가 인증을 획득하였다. 하지만 2014년 해외 유입으로 인한 전국적인 홍역의 집단유행사례가 발생하였고, 여전히 많은 국가의 풍토병 중 하나이기 때문에 주변 국가의 홍역 유행양상에 영향을 받아 매년 산발적인 홍역의 발생이 보고되고 있다 (19). 또한 2019년 전국적인 홍역의 발생이 있었다 (20).
본 연구에서는 2019년 경기도내 홍역 의심환자의 호흡기검체 중 유전자 검출검사 결과 홍역바이러스 양성으로 확인된 98건의 검체를 대상으로 홍역바이러스의 역학적 분포 양상 및 유전학적 분포 양상을 연구하였다. 홍역바이러스의 분자역학적 분석은 지역사회 내 홍역 발생 예방 및 대응을 위한 기초자료로 사용될 수 있고, 또한 홍역바이러스의 유전형 분석을 통해 해외 유입에 대한 증거를 제시할 수 있다.
MATERIALS AND METHODS
검체 수집 및 사례 분류
2019년 1월부터 12월까지 경기도내 홍역 의심환자를 대상으로 채취된 호흡기 검체 중 유전자 검출검사 결과 홍역 양성으로 확인된 98건의 검체를 사용하여 연구를 수행하였다. 98건 검체에 대한 사례 분류를 위해 해외 여행력, 환자 접촉력, 발생 장소 등 역학 정보를 활용하여 집단 발생사례와 산발적 발생사례로 분류하였다.
핵산 추출 및 홍역바이러스 특이유전자 확인
홍역 의심환자의 호흡기도말 검체 140 µL를 QIAamp Viral RNA Mini Kit (QIAGEN, Germany)를 사용하여 제조사에서 제공한 방법에 따라 핵산을 추출하였다.
추출된 핵산을 사용하여 홍역바이러스 특이유전자검출을 위해 실시간 역전사중합효소연쇄반응법(Real-time reverse transcription polymerase chain reaction, real-time RT-PCR)을 질병관리청의 홍역바이러스 유전자 검출검사법에 따라 실시하여 홍역바이러스 특이유전자의 증폭을 확인하였다.
특이유전자 증폭이 확인된 홍역바이러스 핵산의 염기서열분석을 위해 nucleoprotein 유전자의 carboxyl 말단 450 nucleotides를 타겟으로 특이적인 primer를 제작하였고, 역전사중합효소연쇄반응법(Reverse transcription - polymerase chain reaction, RT-PCR)을 실시하였다 (Table 1). RT-PCR은 추출된 핵산 10 µL와 HyQ One step RT-PCR Kit (SNC, Korea)를 사용하여 50℃에서 30분간 reverse transcription 과정을 실시하였고, 94℃에서 2분간 initial PCR activation 과정을 실시한 후 94℃ 30초, 55℃ 30초, 72℃ 30초씩 40회 반복하여 반응시킨 후 72℃에서 10분간 반응을 실시한 후 PCR 반응을 중단하였다 (Table 2).
Table 1.
Primers information for sequencing of N gene
| Primer | Sequence (5’-3’) | Location |
| Forward (MeV216) | TGG AGC TAT GCC ATG GGA GT | 1104-1123 |
| Reverse (MeV214) | TAA CAA TGA TGG AGG GTA GG | 1718-1737 |
Table 2.
Amplification conditions used in N gene RT-PCR
| Step | Temperature | Time | Cycles |
| Reverse transcription | 50℃ | 30 min | 1 |
| Initial PCR activation | 94℃ | 2 min | 1 |
| Denaturation | 94℃ | 30 sec | 40 |
| Annealing | 55℃ | 30 sec | |
| Extention | 72℃ | 30 sec | |
| Final extention | 72℃ | 10 min | 1 |
RT-PCR 과정을 통해 증폭된 634 bp PCR 산물은 QIAxcel DNA High Resolution Kit (QIAGEN)와 QIAxcel (QIAGEN) 자동전기영동장치를 사용하여 확인하였다.
Nucleoprotein 유전자 carboxyl 말단 450 부위의 염기서열 분석 및 유전형 확인
RT-PCR 과정을 통해 증폭이 확인된 634 bp의 PCR 산물은 염기서열분석 반응을 위해 BigDye(R) Terminator v3.1 Cycle Sequencing Kits (Applied Biosystems, USA)와 DNA Engine Tetrad 2 Peltier Thermal Cycler (BIO-RAD, USA)를 사용하여 PCR 반응을 진행하였으며, 반응이 끝나면 ABI 3730xl DNA Analyzer (Applied Biosystems)를 사용하여 염기서열 분석을 실시하였다. 분석된 경기도 발생 홍역바이러스의 염기서열들은 BLAST (Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov/Blast. cgi)를 이용하여 GenBank (https://www.ncbi.nlm. nih.gov/) database에 등록되어 있는 기존에 보고된 염기서열과 비교 분석을 통해 가장 높은 상동성을 나타낸 유전형을 확인하였다. 염기서열들은 MEGA (Molecular Evolutionary Genetics Analysis version 7.0.26) software를 이용하여 clustal W(1.6) method로 서열 정렬 후, neighbor-joining method에 의해 염기서열간의 유전적 거리를 추론하여 phylogenetic tree를 작성하였으며, bootstrap 값은 1,000회의 resampled data로부터 추론하였다.
RESULTS
경기도내 발생 홍역의 역학적 분포 양상
2019년 한 해 동안 경기도에서 홍역 의심환자로 신고된 환자의 호흡기 검체 1,577건을 질병관리청의 홍역바이러스 유전자 검출검사법을 통해 홍역바이러스 특이유전자 증폭을 확인하였을 때 98건의 검체에서 특이유전자 증폭이 확인되었다. 증폭이 확인된 98건의 사례의 역학정보를 기반으로 사례분류를 실시하였고, 세 번의 집단 발생이 있었다. 집단 발생은 각각 Group A, B, C로 명명하였고 집단 발생사례는 총 50건, 산발적 발생사례는 48건으로 분류되어 51%가 집단 발생사례로 분류되었다 (Fig. 1).

Fig. 1
Clasification of measles cases in Gyeonggi province in 2019 based on epidemiological information. Cases of mass infection are named each ‘Group A, B, C’ and individual cases are named ‘Sporadic’.
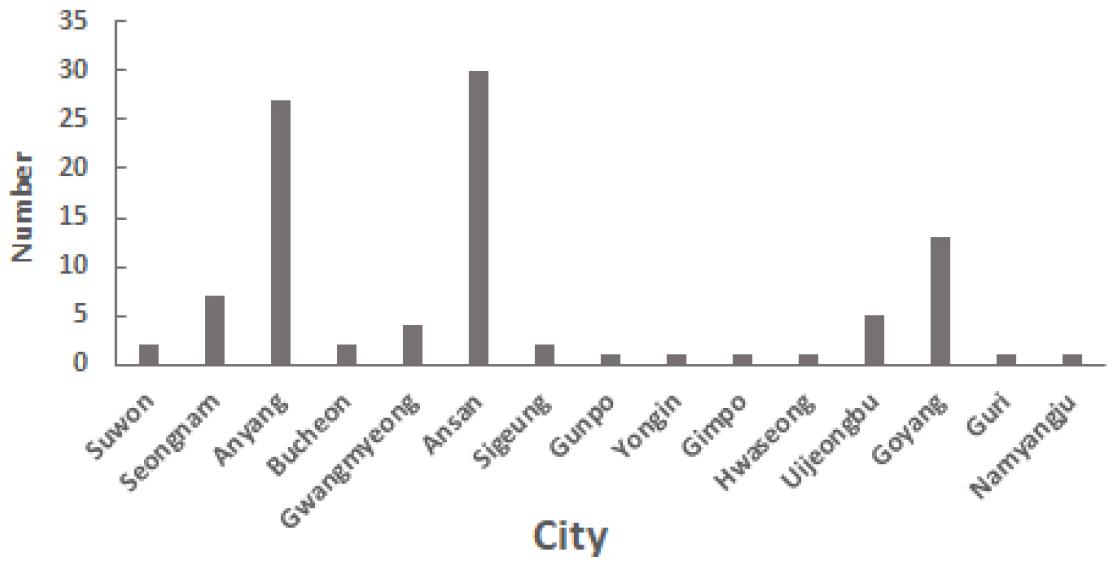
홍역바이러스 특이유전자가 검출된 사례의 시군별 분포를 확인하였을 때, 안산시 30건, 안양시 27건, 고양시 13건 순으로 발생이 많았고, 그 중 집단 발생사례는 안산시, 안양시, 의정부시에서 발생하였다 (Fig. 2). 산발적 발생사례는 수원시, 성남시, 부천시, 광명시 등 15개 시군에서 발생하였다.
월별 홍역바이러스 검출 현황은 Group A의 경우 1월에서 2월에 발생하였고, Group B는 4월, Group C는 2월에 집중 발생하였다 (Fig. 3). 국내 발생 홍역은 주변 국가의 홍역 발생에 영향을 받기 때문에 1월에서 4월 사이에 발생한 88건(90%)은 전 세계적인 홍역 발생 시기의 영향을 받았을 것으로 판단된다 (19).
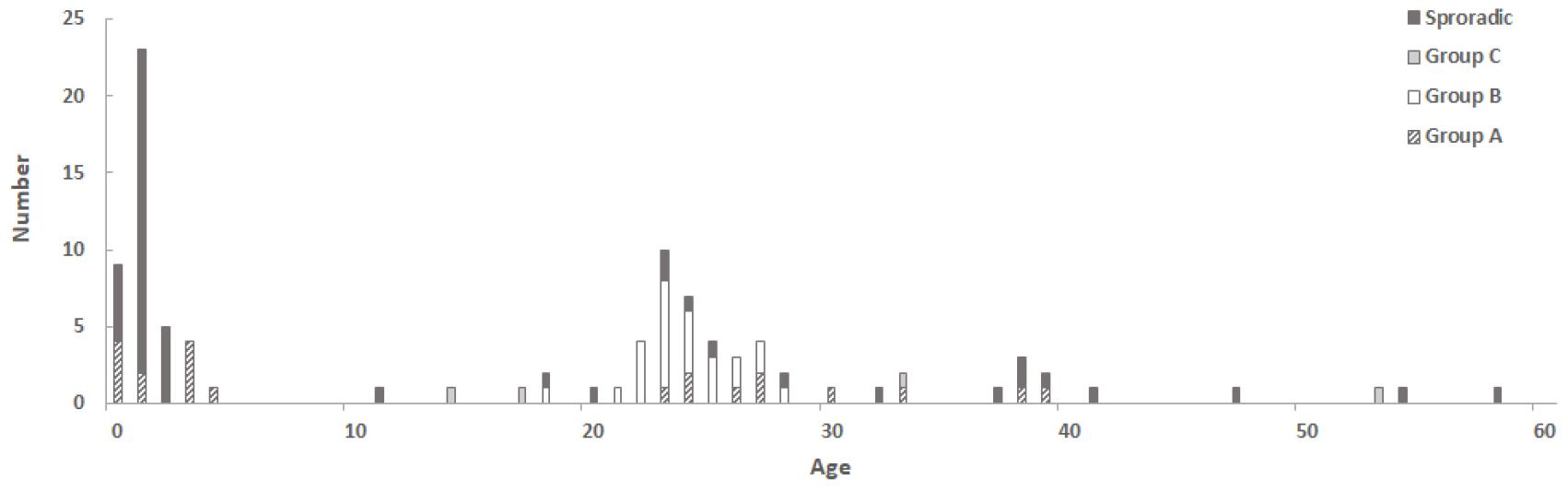
연령별 홍역바이러스 검출 현황은 6세 이하 영유아에서 42건, 20~30대 연령에서 46건 발생하여 80% 이상이 영유아에서 발생하였던 2014년 경기도 홍역 유행사례와는 다른 양상을 보였다 (21). 집단사례별 연령현황은 Group A에서 영유아와 20~30대 연령에서 발생이 많았고, Group B의 경우 20~30대 연령에서 발생이 집중되어 있었다 (Fig. 4). Group C의 경우 연령대의 분포가 다양한 것을 확인할 수 있었다.
경기도내 발생 홍역의 유전학적 분포 양상
홍역바이러스 특이유전자가 검출된 검체 98건을 대상으로 nucleoprotein 유전자의 carboxyl 말단 450 nucleotides 부위를 RT-PCR법을 통해 증폭하였고, 그 중 87건의 검체에서 nucleoprotein 유전자의 증폭이 확인되었다. 증폭되지 않은 11건은 초기에 적은 바이러스의 양으로 유전자 검출검사 양성으로 확인된 후 냉동보관 등의 이유로 소실되었을 것으로 추정된다.
증폭된 nucleoprotein 유전자부위를 염기서열 분석 후 phylogenetic tree 및 BLAST를 통해 유전형을 확인하였다 (Fig. 5). 그 결과 홍역바이러스 D8 유전형이 56건(64.4%)로 가장 많았고, 백신주로 알려진 A 유전형이 18건(20.7%), B3 유전형이 13건(14.9%)으로 확인되었다 (Table 3).
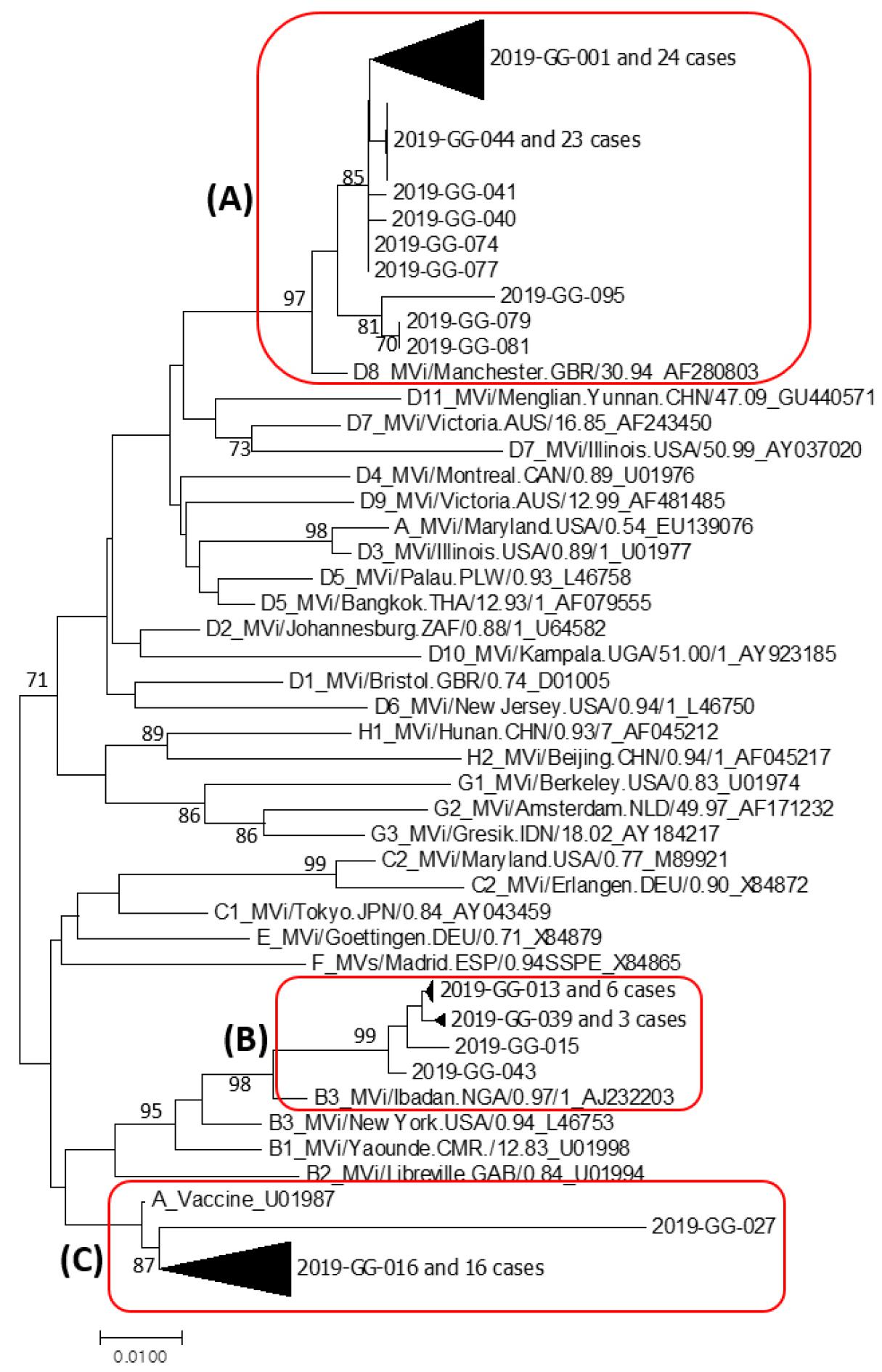
Fig. 5
Phylogenetic tree of measles virus partially N gene. This phylogenetic tree consists of measles virus strains isolated in Gyeonggi province and WHO’s reference strains for measles virus genotyping. Bootstrap values <70% are hidden in the trees. (A) D8 genotype cluster that contains groups A and B, (B) B3 genotypes cluster that contains group C, (C) A genotype cluster.
Table 3.
Genotypes distribution of measles virus isolated in Gyeonggi province in 2019
| Genotype | No. of measles virus (%) |
| A | 18 (21) |
| B3 | 13 (15) |
| D8 | 56 (64) |
| Total | 87 (100) |
역학적 분석을 통해 분류된 집단 사례 Group A, B의 경우 D8 유전형으로 확인되었고, Group C의 경우 B3 유전형으로 확인되었다. D8 유전형으로 분류된 사례를 2018년부터 2019년 사이 보고된 D8 유전형과 비교하였을 때 Group A의 경우 미얀마에서 유행하였던 홍역바이러스와 99.8~100.0% 일치하였고, Group B의 경우 유럽형으로 알려진 이탈리아, 스웨덴, 미국, 베트남 등 14개국 이상의 나라에서 보고된 홍역바이러스와 99.6~99.8% 일치하였다. 산발적 사례의 경우 태국, 미얀마, 미국 등에서 유행한 홍역바이러스와 높은 상동성을 보였다. B3 유전형으로 확인된 Group C의 경우 필리핀에서 유행하였던 홍역바이러스와 99.6~100.0% 일치하였다. 그 외 산발적 사례의 경우 유럽, 말레이시아, 필리핀, 태국, 미얀마에서 유행한 홍역바이러스와 상동성이 높은 것으로 확인되었다 (Table 4).
Table 4.
The similarity of measles virus in Gyeonggi province compared to measles virus reported worldwide during 2018-2019
DISCUSSION
본 연구는 2019년 경기도내 홍역 유전자 검출검사 결과 양성으로 확인된 환자의 호흡기 검체를 대상으로 역학 및 유전형 분석을 실시하였고, 홍역의 유행 양상 및 홍역바이러스의 유전형 분포를 확인하였다. 역학정보 분석 결과 세 건으로 분류된 집단 발생사례는 모두 병원 등 집단시설 내 감염으로 확인되어 2014년 경기도내 홍역 유행사례와 비슷한 양상을 보였다 (21). 이러한 발생 양상을 보았을 때 접촉자 홍역 면역력검사 등 집단시설 내 발생 홍역에 대한 관리가 강화되어야 한다고 판단된다. 그러나 2019년 경기도내 발생 홍역의 유전형분포 및 환자의 연령분포는 2014년 경기도내 발생 홍역과 차이를 보였다. 특히 80% 이상 영유아에서 발생하였던 2014년 발생 홍역과는 달리, 영유아 뿐만 아니라 20~30대 연령대 또한 발생이 많았다. 또한 유전형 분석 결과를 통해 백신주인 A 유전형을 제외한 연령별 홍역바이러스 검출 현황을 확인하였을 때, 영유아에서 20건, 20~30대 연령에서 42건으로, 20~30대에서 2배 이상의 홍역환자가 발생한 것을 확인할 수 있었다. 이는 예방접종 완료 이전의 영유아 외에도 홍역바이러스에 대한 면역력이 없는 연령대가 있음을 시사하며, 1996년에 2차 예방접종이 도입되던 시기에 출생하여 예방접종을 완료하지 못하였거나 예방접종은 완료하였으나 면역감소 등의 이유로 백신 추가접종이 필요한 집단 또한 존재할 것으로 판단된다 (22, 23).
2019년 경기도내 발생 홍역이 국내 발생이 아닌 해외유입 사례임을 확인하기 위해 유전형 분석 결과 D8 및 B3 유전형으로 분류된 사례를 2018년부터 2019년 사이 보고된 해외 홍역사례와 염기서열 비교분석을 수행하였다. 그 결과 미얀마, 유럽, 필리핀 등 해외에서 유행한 홍역사례와 99.6~100.0% 일치하여 상동성이 높은 것을 확인하였다. 이는 2019년 경기도내 발생 홍역이 해외유입 홍역이라는 계통분류학적 증거가 된다. 역학정보 분석 결과 경기도내 발생 홍역의 최초 감염원은 확인되지 않아 홍역의 국내 유입 경로는 불분명하지만, 염기서열 비교분석 결과를 근거로 최초 감염원은 해외유입환자이며, 국내 유입 후 지역사회 내 2차 전파가 되었을 것으로 추정할 수 있다.
WHO는 코로나바이러스감염증-19의 대유행으로 홍역에 대한 감시 및 예방접종률이 전세계적으로 감소했다고 보고하였다 (24). 우리나라는 국민예방접종사업을 통해 홍역 예방백신의 접종률이 96% 이상 유지되고 있지만 (25), 해외 유입에 의한 홍역 발생 가능성이 존재하기 때문에 본 연구 결과는 해외 유입 홍역에 대한 사례 보고 결과로써 향후 국내 홍역 집단 발생 시 사례 비교 등 분석을 위한 자료로 활용될 수 있으며, 연구에서 사용된 홍역바이러스의 유전형 분석법 및 계통분류방법은 최초 감염원이 불분명한 홍역 사례에 대해 국내 유입 경로를 분자역학적으로 추적할 수 있는 방법을 제시한다. 또한, 역학적 분석 결과는 고위험집단에 대한 면역력 검사 실시 등 홍역의 예방을 위한 방역체계 구축에 중요한 자료로 활용될 것으로 판단된다.